Caracterización Molecular y Análisis del Rango de Hospedadores de Bacteriófagos Líticos Dirigidos a Pseudomonas aeruginosa Multirresistente Aislados de Aaguas Residuales en Perú
Molecular Characterization and Host Range Analysis of Lytic Bacteriophages Targeting Multidrug-Resistant Pseudomonas aeruginosa from Wastewater in PeruJosé Guillermo González Cabeza 1*, José Luis Huayán Radas 2,Ofelia Magdalena Córdova Paz Soldán 3, Pedro Salvador González Jara 4,
Franklin Roger Vargas Vásquez 5, Jorge Luis Leiva Piedra 6
Franklin Roger Vargas Vásquez 5, Jorge Luis Leiva Piedra 6
1Universidad Privada Antenor Orrego / Trujillo / Perú; jgonzalezc1@upao.edu.pe.
2Universidad Nacional de Trujillo/Trujillo/Perú; josehuayanr@gmail.com.
3Universidad Privada Antenor Orrego / Trujillo / Perú; ocordovap@upao.edu.pe.
4Universidad Privada Antenor Orrego / Trujillo / Perú; pgonzalezj2@upao.edu.pe.
5Universidad Nacional de Trujillo/Trujillo/Perú; frvargasv@yahoo.es.
6Universidad Tecnológica del Perú/Trujillo/Perú; jleiva@utp.edu.pe.
* Correspondence: jgonzalezc1@upao.edu.pe; Tel.: (+51 959 662 769).
RESUMEN
La Pseudomonas aeruginosa multirresistente (PAMR) constituye un importante problema de salud pública a nivel mundial, que limita las opciones terapéuticas y aumenta la morbilidad y mortalidad. Este estudio presenta el aislamiento, caracterización molecular y evaluación de la estabilidad de bacteriófagos líticos activos contra cepas de PAMR recuperadas de aguas residuales urbanas en Trujillo, Perú. Se confirmaron dieciséis aislamientos clínicos de PAMR mediante pruebas fenotípicas y bioquímicas, y se evaluó su resistencia frente a 13 antibióticos. Se obtuvieron ocho bacteriófagos líticos, que fueron evaluados en cuanto a viabilidad bajo diferentes temperaturas de almacenamiento (4°C, −30°C, −80°C), temperaturas elevadas (37–47°C), condiciones de pH (3, 7, 9), exposición a radiación UV (5–60 s) y rango de hospedero frente a todos los aislados bacterianos. La caracterización molecular se realizó mediante polimorfismo de longitud de fragmentos de restricción (RFLP) utilizando las enzimas EcoRI, BamHI y HindIII. Los fagos F1AO-510 y F1AO-506 mostraron el rango de hospedero más amplio, lisando el 56,25% y el 43,75% de las cepas de PAMR, respectivamente. La mayoría de los fagos mantuvo su infectividad durante tres meses a 4°C y permaneció estable a −80°C, mientras que exposiciones a UV superiores a 30 segundos redujeron notablemente su viabilidad. Los perfiles RFLP revelaron patrones genómicos diferenciados, evidenciando su diversidad genética y su potencial para integrar cócteles fágicos específicos. Estos hallazgos resaltan el valor terapéutico de los bacteriófagos líticos como agentes antimicrobianos alternativos y su aplicabilidad en intervenciones fágicas en regiones con alta prevalencia de resistencia antimicrobiana.
Palabras clave: Pseudomonas aeruginosa, multirresistencia, terapia con bacteriófagos, microbiología de aguas residuales, estabilidad fágica, rango de hospedero, análisis RFLP.
ABSTRACT
Multidrug-resistant Pseudomonas aeruginosa (MDR-PA) represents a major global public health problem, limiting therapeutic options and increasing morbidity and mortality. This study reports the isolation, molecular characterization, and stability assessment of lytic bacteriophages active against MDR-PA strains recovered from urban wastewater in Trujillo, Peru. Sixteen clinical MDR-PA isolates were confirmed through phenotypic and biochemical tests, and their resistance to 13 antibiotics was evaluated. Eight lytic bacteriophages were obtained and assessed for viability under different storage temperatures (4°C, −30°C, −80°C), elevated temperatures (37–47°C), pH conditions (3, 7, 9), UV radiation exposure (5–60 s), and host range against all bacterial isolates. Molecular characterization was performed by restriction fragment length polymorphism (RFLP) analysis using EcoRI, BamHI, and HindIII enzymes. Phages F1AO-510 and F1AO-506 exhibited the broadest host ranges, lysing 56.25% and 43.75% of MDR-PA strains, respectively. Most phages retained infectivity for three months at 4°C and remained stable at −80°C, whereas UV exposures longer than 30 seconds markedly reduced viability. RFLP profiles revealed distinct genomic patterns, highlighting their genetic diversity and potential for inclusion in targeted phage cocktails. These findings underscore the therapeutic value of lytic bacteriophages as alternative antimicrobial agents and their applicability in phage-based interventions in regions with a high prevalence of antimicrobial resistance.
Keywords: Pseudomonas aeruginosa, multidrug resistance, bacteriophage therapy, wastewater microbiology, phage stability, host range, RFLP analysis.
INTRODUCCIÓN
La resistencia a los antimicrobianos (RAM) se ha convertido en una de las mayores amenazas para la salud pública a nivel global, impulsada por el uso indiscriminado de antibióticos y la falta de políticas efectivas de control 1,2. Este fenómeno no discrimina entre países desarrollados y en desarrollo: mientras en la India se registran 214,000 muertes anuales de recién nacidos por sepsis, en Estados Unidos y Europa las cifras alcanzan 23,000 y 25,000 defunciones, respectivamente 3. Las proyecciones son aún más alarmantes, sugiriendo que para 2050 las bacterias multirresistentes podrían causar 10 millones de muertes anuales, superando el impacto de la pandemia de COVID-19 4. Entre los patógenos prioritarios, Pseudomonas aeruginosa destaca por su capacidad para causar infecciones nosocomiales y comunitarias graves 5,6. Su ubicuidad en ambientes húmedos y su habilidad para formar biopelículas lo convierten en un patógeno difícil de erradicar 7, 8. A nivel nosocomial, ocasiona infecciones en casi todas las partes del cuerpo humano, principalmente en áreas húmedas, llegando a colonizar cualquier sitio expuesto 9. La resistencia intrínseca de este microorganismo, mediada por su membrana externa impermeable y sistemas de eflujo activo 10, limita las opciones terapéuticas, especialmente en pacientes inmunocomprometidos 11. Esta situación llevó a la OMS y los CDC a clasificar a P. aeruginosa multirresistente (PAMR) como una amenaza crítica para la salud global 12.
Los mecanismos de resistencia en PAMR son diversos e incluyen la impermeabilidad de la membrana, la activación de bombas de eflujo y la producción de β-lactamasas 13, 14. Además, la transferencia horizontal de genes a través de elementos móviles como plásmidos y transposones acelera la diseminación de resistencia 15, 16. Frente a este escenario, los bacteriófagos emergen como una alternativa prometedora debido a su especificidad, seguridad y capacidad para lisar bacterias resistentes 17, 18. Estos virus, inocuos para humanos y animales, han demostrado eficacia en reducir cargas bacterianas en modelos experimentales 19,20, y su diversidad genética (genomas de 15–500 kb) los hace candidatos ideales para terapias personalizadas 21, 22.
El creciente interés en la fagoterapia, ha tomado gran interés, debido a la rápida adaptación de bacterias multirresistentes a antimicrobianos, sustentándose en resultados alentadores de ensayos clínicos que muestran su potencial para tratar infecciones por PAMR 23,24. A lo largo de los años, diversos estudios (modelos experimentales en animales o ensayos clínicos en humanos) enfocados en la terapia con bacteriófagos de P. aeruginosa, para el tratamiento de septicemias en diferentes infecciones han arrojado resultados alentadores, ya sea, en la disminución del recuento bacteriano, evolución de la enfermedad, reducción de la frecuencia de infecciones nosocomiales y/o descenso de la mortalidad 25. Sin embargo, su aplicación requiere una caracterización exhaustiva que incluya estabilidad fisicoquímica, rango de hospedero y análisis molecular. Los altos índices de PAMR a antimicrobianos de última generación, cada vez son más frecuente; lo que supone una seria amenaza a la terapia clínica; por ello, el presente estudio buscó realizar una caracterización molecular por RFLPs de bacteriófagos líticos de P. aeruginosa multirresistentes (PAMR), aislados de agua de albañal de Centros de Salud y pozas de oxidación, y brinda una propuesta de fagoterapia para infecciones producidas por PAMR.
MATERIALES AND METODOS
Muestreo y procesamiento inicial
Recolección de muestras:
• Se recolectaron 300 mL de agua residual de centros de salud y pozas de oxidación primarias/secundarias del distrito de Chao (La Libertad, Perú), en botellas estériles de boca ancha.
• Las muestras se transportaron en cooler a 4°C y se almacenaron a -80°C en el Laboratorio de Microbiología Molecular y Biotecnología de la UPAO 26.
Cepas bacterianas:
• Se utilizaron 16 cepas clínicas de P. aeruginosa multirresistente (PAMR), previamente identificadas por sistemas automatizados en hospitales de Trujillo.
• Controles: Se incluyó la cepa ATCC 27853 como control positivo en todas las pruebas.
Aislamiento y purificación de bacteriófagos
Obtención de lisados fágicos:
A. Enriquecimiento:
• Se incubaron 10 mL de muestra con 90 mL de buffer SM + 5 mL de caldo LB 2X + 1 mL de cultivo ON de PAMR + 200 µL de CaCl₂ (37°C, 24 h).
• Se añadió cloroformo (4% v/v), se centrifugó (4,000 rpm, 15 min), y se recuperó el sobrenadante (lisado crudo) 26, 27.
B. Detección preliminar (Spot test):
• Se sembraron 100 µL de cultivo ON de PAMR en top agar (con CaCl₂ 10 mM) sobre placas de agar nutritivo.
• Se agregaron 10 µL de lisado crudo y se incubó (37°C, 24 h). La presencia de halos de lisis confirmó actividad fágica 28.
C. Purificación:
• Se realizaron diluciones seriadas (10⁻³ a 10⁻⁹) del lisado crudo en buffer SM.
• Se seleccionaron placas con halos de lisis aislados y bien definidos. El proceso se repitió 5 veces para obtener fagos puros 29.
D. Cuantificación:
• Se calcularon unidades formadoras de placa (UFP/mL) mediante diluciones seriadas (10⁻⁷ a 10⁻¹⁰) y técnica de doble capa.
Caracterización fisicoquímica de bacteriófagos
Estabilidad a temperaturas:
• Almacenamiento: 1 mL de cada fago purificado se almacenó a -30°C y -80°C (6 réplicas por condición). La infectividad se evaluó a los 30 días mediante Spot test.
• Termoestabilidad: Se incubaron suspensiones fágicas a 37°C, 42°C y 47°C (1 h, agitación). Se midió la infectividad residual 30.
Resistencia a UV:
• Se expusieron 200 µL de fagos a UV-C (254 nm, 30 cm) durante 5, 30 y 60 segundos (6 réplicas). La viabilidad se evaluó con diluciones seriadas.
Rango de hospedero:
• Se enfrentaron los fagos purificados a las 16 cepas PAMR mediante Spot test. Se registró el porcentaje de cepas susceptibles.
Estabilidad a pH:
• Se incubaron 100 µL de fagos en buffer SM (pH 3, 7 y 10; 37°C, 1 h). La infectividad se evaluó post-incubación 27.
Análisis molecular
Extracción de DNA fágico y bacteriano:
• DNA fágico: Se purificó con el kit GeneJET Genomic DNA (Thermo Scientific). Se cuantificó con Nanodrop (≥100 ng/µL).
• DNA bacteriano: Se extrajo mediante lisis térmica (100°C, 15 min) y se confirmó por PCR del gen O-acetilasa (232 pb) 31.
Digestión con enzimas de restricción (RFLPs):
• Selección de fagos: Solo se analizaron los 2 fagos con mayor actividad lítica (F1AO-510 y F1AO-506).
• Protocolo: Se digirieron 10 µL de DNA con EcoRI, BamHI y HindIII (según protocolo del fabricante). Los fragmentos se visualizaron en geles de agarosa al 1% (70 V, 40 min).
Controles experimentales:
• Negativos: Buffer SM estéril en lugar de DNA fágico.
• Positivos: DNA de fagos estándar con patrones de restricción conocidos.
RESULTADOS
Obtención y caracterización de las cepas de P aeruginosa

Figura 1. Observación Macroscópica de las 8 cepas de P aeruginosa multirresistentes (PAMR) seleccionadas, en agar cetrimide: AO-502(A), AO-506(B), AO-508(C), AO-509(D), AO-510(E), AO-513(F), AO-514(G), AO-515(H); aisladas de muestras biológicas recolectadas de centros de salud y laboratorios clínicos de Trujillo.
En la Figura 1 se muestran los ocho aislados de PAMR en agar cetrimide: AO-502, AO-506, AO-508, AO-509, AO-510, AO-513, AO-514 y AO-515; en el cual se observa, la presencia de colonias con diversos morfotipos descritos para Pseudomonas aeruginosa (colonias de aspecto seco, metálico y mucoide), los cuales muchas veces están asociados a virulência. Asimismo, se realizó la comprobación fenotípica mediante pruebas bioquímicas y de susceptibilidad antimicrobiana convencionales, empleando diversos medios de cultivos corroborándose que las muestras biológicas erán al idóneas para iniciar el trabajo, tal como aparecen descritas en la Tabla 1 Material Suplementario, las cuales coinciden con las características culturales y fisiológicas propias de Pseudomonas aeruginosa, el ser bacilos Gram negativos, no fermentadores (TSI), productor de pigmentos (piocianina/pioverdina en agar cetrimide). En la Tabla 2 Material Suplementario se muestran los resultados de los patrones de sensibilidad in vitro de los 16 aislados PAMR, frente a 13 antimicrobianos propuestos, confirmándose la multirresistencia de los aislados, a excepción de la AO-504 que solo mostro resistencia frente a dos familias de antimicrobianos.

Figura 2. Multiresistencia de cepa de PAMR AO-510, mediante su perfil de sensibilidad por técnica de difusión de disco (Kirby-Bauer) frente a 12 antimicrobianos: ceftazidime (CAZ), colistin sulphate (CT), ciprofloxacin (CIP), amikacina (AK), tobramycin (TOB), aztreonam (ATM), gentamicina (CN), piperacilin/tazobactam (TZP), imipenem (IPM), piperaciclin (PRL), cefepime (FEP), oxfloxacin (OFX).

Figura 3. Curva de crecimiento: Inicio de la Fase exponencial “Log” a las 4 horas (flecha verde) de la cepa control de P. aeruginosa ATCC 27853, correlacionando la densidad óptica (D.O) versus el tiempo de crecimiento (horas).
Spot Test

Figura 4. Confirmación de ocho lisado fágicos para los aislados de PAMR (AO-502, 506, 508 – 510, 513 – 515), mediante la observación de una placa lítica centra - prueba de la gota “spot test”.
En la obtención de lisados fágicos para cada una de las PAMR, se hallaron ocho lisados que mostraron actividad lítica sobre las cepas AO-502, AO-506, AO-508, AO-509, AO-510, AO-513, AO-514 y AO-515, (Figura 4) por una calva de lisis central mediante la prueba de la gota “Spot test”.
Infectividad frente a almacenamiento a bajas temperaturas

Figura 5. Prueba de comparación de medias TUKEY. (a) para el efecto de los meses de conservación y (b) comportamiento de las cepas de phagos frente a los meses de conservación

Figura 6. Prueba de comparación de medias TUKEY. (a) para el efecto de la conservación a 4°C, -30°C y -80°C y (b) comportamiento de las cepas de phagos frente a la temperatura de conservación.
Los resultados obtenidos en las pruebas de infectividad de las cepas de phagos frente a los meses y temperaturas de almacenamiento, nos muestran que, los meses de conservación no tuvieron un efecto sobre este parámetro, evidenciándose esto en el p-valor = 0.378 (figura 5A), sin embargo, en el comportamiento de las cepas de phagos, podemos observar que, entre ellas, la cepa que mostró un mejor comportamiento fue la cepa FA506, quien arrojó el valor promedio más alto con 4x10E9, pero el p-valor = 0.185, nos dice que esta diferencia no fue estadísticamente significativa (figura 5B). Asimismo, en la prueba de temperatura de conservación (figura 6A), se observó que esta tampoco tuvo un efecto sobre la infectividad de los phagos, evidenciándose que no existió un efecto significativo a nivel estadístico, corroborado esto por el p-valor = 0.300, sin embargo, se nota una ligera diferencia entre los valores promedios, resaltando la temperatura de 04°C. Por otro lado, en la respuesta de las cepas, la cepa FA0506, mostró el valor promedio de titulación más alto, sin embargo, el p-valor = 0.477, nos dice que no fue significativo a nivel estadístico (figura 6B); lo que nos permite inferir que tanto el tiempo (0, 3 y 6 meses) como la temperatura (04°C, -30°C y -80°C), no afectaron la infectividad de las cepas de phagos.
Infectividad frente a radiación UV

Figura 7. Prueba de comparación de medias TUKEY. (a) para del tiempo de la Radiación UV en la estabilidad de los bacteriófagos y (b) para el comportamiento de las cepas de phagos frente a la UV.
En la prueba de infectividad de las cepas de phagos frente a diferentes tiempos de exposición de luz UV, podemos observar que, este factor no tuvo un efecto sobre este parámetro, a pesar de que se observa una variación entre los valores promedios de 5, 30 y 60 seg. de exposición frente a 0 seg, sin embargo, el p-valor = 0.221, nos dice que esta no fue significativa a nivel estadístico (figura 7A). Por otro lado, en la respuesta de las cepas, se observa que la cepa FA0506, arrojó el valor de titulación más alto, pero el p-valor = 0.307, nos refiere que, a pesar de estar en un subconjunto diferente, este no fue estadísticamente significativo (figura 7B). Por lo que podemos inferir que los diferentes tiempos de exposición de UV, en esta prueba, no afectaron la capacidad infectiva de las cepas de phagos estudiadas.
Termo estabilidad

Figura 8. Prueba de comparación de medias TUKEY. (a) para el efecto de las altas temperaturas en la estabilidad de los bacteriófagos y (b) para el comportamiento de las cepas de phagos en estudio.
Los resultados obtenidos en la prueba de termoestabilidad de las cepas de phagos, podemos observar que las temperaturas a las que fueron sometidos no tuvieron un efecto sobre su nivel de infectividad, ya que en la prueba de comparación de medias el p-valor obtenido fue de 0.229, lo que denota que la diferencia entre estos no fue estadísticamente significativa (figura 8A), a pesar de que la temperatura de 4°C arrojó el valor promedio de titulación más alto. Por otro lado, en el comportamiento de las cepas, la cepa que arrojó el valor promedio de titulación fue la FA0506, sin embargo, a pesar de ello, el p-valor = 0.285, nos dice que no hubo una diferencia significativa a nivel estadístico entre ellos (figura 8B). Esto nos permite inferir que las diferentes temperaturas a las que fueron sometidas las cepas de phagos en estudio, no afectaron su nivel de infectividad.
Estabilidad a diferentes pH
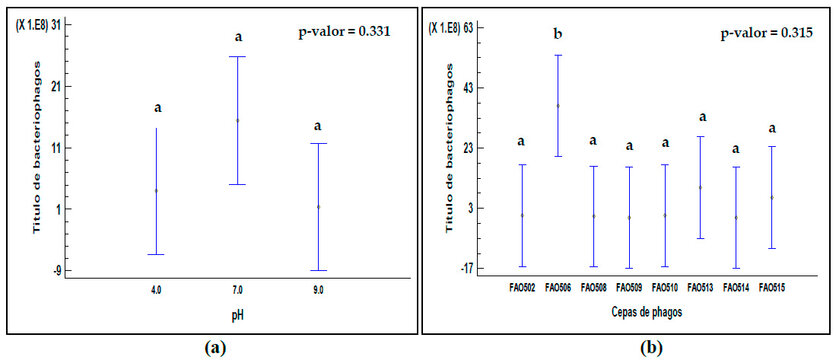
Figura 9. Prueba de comparación de medias TUKEY. (a) para el efecto del pH en la estabilidad de los bacteriófagos y (b) comportamiento de las cepas de phagos en estudio frente a los diversos pH.
Respecto a los efectos de los pH 3, 7 y 9 durante una hora a 37ºC sobre la infección lítica de los fagos, podemos observar en la prueba de comparación de medias (figura 9A) que estas no tuvieron un efecto sobre este comportamiento, ya que el p-valor = 0.331, nos dice que, entre ellas, no existió una diferencia significativa a nivel estadístico. Asimismo, en la prueba de comparación de medias, para determinar el comportamiento de las cepas, podemos observar que la cepa FA0506, arrojó los valores de titulación más altos, sin embargo, el p-valor = 0.315, nos dice que, en dicho parámetro, no hubo una diferencia significativa a nivel estadístico. Estos resultados nos permiten inferir que el pH no influye ni afecta la capacidad de infección de los phagos en estudio.
Rango de hospedero

Tabla 3. Cuantificación de bacteriófagos (UFP/mL) de las suspensiones madre (50 mL) de 8 bacteriófagos aislados
En la tabla 3 se presentan los títulos alcanzados (UFP/mL) de los 8 bacteriófagos candidatos, la codificación del bacteriófago se estableció de la siguiente manera: F (bacteriófago), AO (Antenor Orrego) y número del aislado bacteriano de procedencia. Mostrando mayor título el F1AO-506, con 1010 UFC/mL.

Tabla 4. Rango de hospedero de 8 bacteriófagos frente a 16 cepas PAMR, según su porcentaje de lisis.
En la Tabla 4 se observan los resultados correspondientes a la determinación del rango de hospedero de los 8 bacteriófagos aislados y purificados frente a las 16 cepas de PAMR, resaltando la actividad lítica presentada por los bacteriófagos F1AO-510 y F1AO-506, que tuvieron actividad lítica sobre 9 (56,25%) y 7 (43.75%) de los 16 aislados de PAMR evaluadas. Asimismo, se observa que los aislados PAMR más vulnerable a la acción fágica fueron la AO-501 y AO-505, que fueron infectadas por un 75% de los bacteriófagos aislados, mientras que las más resistentes a la infección fueron la AO-502, AO-503, AO-508, AO-513, AO-515 y AO-516 que solo fueron infectadas por sus correspondientes bacteriófagos aislados (12,5%).
Obtención del DNA fágico y análisis RFLPs

Figura 10. Patrón de restricción por corrido electroforético del DNA del bacteriófago F1A0-510 con EcoRI, mediante la técnica RFLPs, visto en gel de agarosa al 0.7% TAE y visualizado con Bromuro de etidio 0.01%, nótese la migración de moléculas.

Figura 11. Corrido electroforético del DNA no digerido de los bacteriófago F1AO-510 y F1AO-513, con la enzima BamHI, mediante la técnica RFLPs, visto en gel de agarosa al 0.7% TAE y visualizado con Bromuro de etidio 0.01%.
En la Figura 10 y 11, se muestra el DNA digerido (migración de moléculas) del mejor candidato seleccionado F1A0-510 cortado con EcoRI y no digerido de F1AO-510 y F1AO-513, con la enzima BamHI, mediante el patrón de restricción en corrido electroforético (técnica RFLPs). Evidenciándose en estos últimos, que no hay migración de moléculas.
Identificación por PCR de las cepas de PAMR aisladas

Figura 12. Amplificación del amplicón O - acetilasa de P. aeruginosa mediante PCR, para la identificación molecular de las PAMR aisladas AO-501 – 516 (carriles del 1 – 16), cepa ATCC 27853 de P. aeruginosa como control positivo (C+).
En la Figura 12, se observa las bandas de aproximadamente 232 pb del amplicón O – acetilasa (OatA) de P. aeruginosa mediante PCR-FRLPs, para la identificación molecular de las PAMR aisladas AO-501 – 516. Donde se usó la cepa ATCC 27853 de P. aeruginosa como control positivo. Y se contrastaron todas las muestras con el marcador de peso molecular de 1 Kb (M). Determinándose que las 16 cepas aisladas pertenecen al género de P. aeruginosa.
DISCUSIÓN
Obtención y caracterización de las cepas de P aeruginosa multirresistentes (PAMR)
La caracterización fenotípica y microscópica de la cepa control ATCC 2785316 y las 16 cepas reactivadas (AO-501 – 515), mostraron que todas eran bacilos Gram (-). En sus características macroscópicas, y su bioquímica, se observaron colonias circulares, convexas, brillantes, húmedas y secas, de bordes regulares y con formación de pigmentos (piocianina y pioverdina) en agar cetrimide (Figura 1) y cepas alcalinas (ALK) en medio TSI 32, lo que indico estar frente a P. aeruginosa (Tabla 1 Material Suplementario). Por otro lado, los resultados del perfil de sensibilidad in vitro (método de difusión con discos de Kirby-Bauer) (Tabla 2 Material Suplementario), cuya interpretación de los resultados se realizó de acuerdo a los parámetros establecidos por el manual M100 CLSI 2021 (Clinical and Laboratory Standards Institute) usando como cepa de control P. aeruginosa ATCC 27853, permitió determinar la multiresistencia (PAMR) de 10 de las 16 cepas aisladas, en base al diámetro (milímetros) del halo de inhibición del crecimiento mostrado frente a 13 antimicrobianos enfrentados (Tabla 2 Material Suplementario).
La resistencia natural de P. aeruginosa se debe principalmente a la baja permeabilidad de la membrana externa, la expresión de bombas de eflujo y la producción de β-lactamasas inducibles por AMPc; sin embargo, su alta adaptabilidad debido a factores genéticos, incluyendo hipermutabilidad, le permite desarrollar fácilmente virulencia y resistencia 33. En el antibiograma de la cepa de PAMR AO-510, mediante su perfil de sensibilidad frente a 12 antimicrobianos (Figura 2), se determinó que la cepa seleccionada es de resistencia intermedia para aztreonam (ATM) y piperacilin/tazobactam (TZP) y resistente para los nueve antimicrobianos restantes. La resistencia a imipenem, meropenem y cefalosporinas posiblemente se deba a la adquisición de β-lactamasas transferibles o la impermeabilidad de la membrana externa (alteración de la porina OprD), la cual también genera resistencia a aminoglucósidos 34. Así mismo, la sobreproducción de sus bombas de eflujo MexAB-OprM y MexXY-OprM pueden desencadenar resistencia a las quinolonas, macrólidos, aminoglicósidos, sulfonamidas, tetraciclinas y muchos β-lactámicos 35.
Spot test
La presencia de fagos de PAMR (cepas F1AO-502, 506, 508 – 510, 513 – 515), fue confirmada por la formación de una placa lítica central, mediante la prueba de la gota “Spot test” 28 (Figura 4). Los bacteriófagos fueron aislados mediante la técnica de 26, 27, estos provinieron de muestras de agua de albañal (parte líquida y sedimento) procedentes de centros de salud y pozas de oxidación primarias y secundarias de Chao (Anexo 6 y 7) ya que, en estas aguas se encuentran asociadas elevadas cargas fágicas; que constituyen un control biológico para las colonias bacterianas 36, 25.
Infectividad frente a almacenamiento a bajas temperatura
La conservación de los bacteriófagos a bajas temperaturas, por periodos prolongados, facilitar su disponibilidad para futuras investigaciones o aplicaciones terapéuticas; por ello, se buscó determinar su infectividad, conservados a bajas temperaturas, durante 3 a 6 meses (Figura 6), observándose que las cepas son resistentes y tolerantes a la conservación; a excepción de la cepa F1AO-509, quién a los - 30 y - 80 °C mostró ser sensible, disminuyendo hasta 5 órdenes logarítmicas (-83.3%), manteniendo una efectividad solo del 16.6%, siendo las cepas resistentes F1AO-508 y F1AO-515 quienes mantuvieron un 87.5 y 88.8% de infectividad respectivamente. A ambas temperaturas de conservación, las cepas restantes mostraron ser tolerante con un porcentaje de infectividad por encima del 50%. Estos resultados contrastan con los de Luciano y Jonás (2017) 37, quienes en el bacteriófago ФGF1 de E. coli, expuesto a temperaturas de -20 ºC por 4 meses; determinando que esta es la temperatura óptima de almacenamiento, ya que después de este tiempo, mantuvo su infectividad por encima del 50%. Los resultados de la conservación a bajas temperaturas fueron contrastados con una conservación a 4 °C; ya que, durante el trabajo de esta caracterización fenotípica, se determinó que esta temperatura de refrigeración es favorable para la conservación de bacteriófagos. Así, se evaluó el efecto de la conservación a 4 °C a los tres y seis meses (Figura 5), observándose buenos resultados. Todas las cepas mostraron ser resistentes, resaltando los bacteriófagos F1AO-510 y F1AO-515 que conservaron un 100% de su infectividad y F1AO-506, con un 90% de infectividad; todas las demás cepas se mantuvieron por encima del 75% de infectividad.
Infectividad frente a radiación UV
La supervivencia de un bacteriófago frente a la radiación UV depende de su capacidad para utilizar los sistemas de reparación del ADN que poseen las células hospederas o por una enzima que está codificada en su genoma y está comprometida en los mecanismos de reparación oscura 38. En la determinación del efecto de la luz UV germicida (UV-C, 254 nm) sobre la infectividad de los bacteriófagos (Figura 7), se observó que, a los 5 minutos de exposición, las cepas F1AO-508 y F1AO-515, mostraron ser tolerantes, ya que solo disminuyeron en dos y tres órdenes logarítmicas, manteniendo una infectividad del 62.5 y 77.7 % respectivamente; mientras que, el resto de cepas mostraron ser resistentes, pues su disminución máxima fue de un orden logarítmico; siendo las cepas F1AO-509 y F1AO-510, las más resistentes, pues mantuvieron su infectividad del 100 %. A partir de los 30 segundos de exposición, solo la cepa F1AO-509 mostró ser resistente, ya que tan solo disminuyó un orden logarítmico (infectividad del 83.3%). Resultados similares se observaron con el bacteriófago uB-19 de Bacillus thuringiensis, que mantuvo su infectividad de un 70% a los 40 segundos de haber sido expuesto a UV-C 30. Todas las cepas restantes, a partir de los 30 y hasta los 60 segundos de exposición, mostraron ser sensibles al efecto del UV-C, pues disminuyeron más de 4 órdenes logarítmicas. Es decir, disminuyeron más del 70% de su infectividad inicial, resaltando la cepa F1AO-514, que perdió su infectividad en un 100%; esta disminución total de la infectividad por exposición a la luz ultravioleta contrasta con los resultados obtenidos por 39, donde se redujo la infectividad del bacteriófago Vf1 de Vibrio fluvialis, en un 100% a los 90 segundos de exposición.
Termo estabilidad
Algunos bacteriófagos presentan envoltura lipídica que se deriva de la célula hospedera. La conformación y organización proteica de sus cápsides les confiere la habilidad de permanecer viables por largos períodos de tiempo en condiciones adversas, como la exposición a ADN-asas, variaciones de pH y temperatura 40. La determinación de la infectividad de los bacteriófagos, expuestos durante una hora a elevadas temperaturas (Figura 7); mostró que, a 37 °C las cepas F1AO-509 y F1AO-515 son termotolerantes, con una disminución de dos órdenes logarítmicas; mientras que las cepas restantes mostraron ser resistentes, ya que su disminución máxima, fue de un orden logarítmico. A los 42 y 47 °C, las cepas mostraron ser resistentes y tolerantes, con excepción del bacteriófago F1AO-509, quien mostró ser termosensible, ya que disminuyó hasta un 83.3% su infectividad; reduciéndose hasta en cinco órdenes logarítmicas; estos resultados son comparables con los obtenidos por Dominick et al, 39, en donde el bacteriófago Vf1 de Vibrio fluvialis, perdió más del 80 % de su infectividad a la temperatura de 40 ºC. Los fagos resistentes F1AO-508 y F1AO-510 mantuvieron una infectividad de 87.5 %, así mismo resalta el fago F1AO-514 que mantuvo un 100% de infectividad frente a los tres tratamientos térmicos. Todas las cepas restantes mostraron ser termotolerantes a 47°C. Esta termoestabilidad la evidenciaron García y Porras (2018) 25,quienes en los bacteriófagos LC1, LC3, LR2 y LR3 de P. aeruginosa, observaron que más del 50 % de su infectividad se mantuvo hasta temperaturas de 50 ºC; decayendo drásticamente al ser expuestos a temperaturas superiores a los 60 ºC, perdiendo desde el 70 al 100 % de su infectividad. Esta capacidad de los fagos de soportar las temperaturas fisiológicas en los organismos en los cuales serán inoculados es un criterio importante al momento de su selección, para su uso terapéutico.
Estabilidad a diferentes Ph
Las cepas a pH ácido y alcalino mostraron ser resistentes y tolerantes, con excepción del bacteriófago F1AO-508, el cual, a pH 4, disminuyó 7 unidades logarítmicas (-87.5%) manteniendo tan solo un 12.5% de su infectividad; mientras que a pH 9, demostró ser tolerante, disminuyendo solo 2 unidades logarítmicas (-25%), manteniendo un 75% de su infectividad. Resultados semejantes a los obtenidos por Guo et al (2019) 41, quienes determinaron la resistencia del bacteriófago vB_PaeM_SCUT-S2 de P. aeruginosa, el cual también mostró resistencia a la alcalinidad, ya que mantuvo un 100 % de infectividad, sin embargo, mostró alta sensibilidad a un pH 3, ya que perdió más del 60 % de su infectividad. De las cepas resistentes a pH ácido, resaltan F1AO-502, F1AO-509, F1AO-510, F1AO-513 y F1AO-515 que mantuvieron el 100% de su infectividad; de estos, solo la cepa F1AO-513 mantuvo un 100% de su infectividad a pH alcalino, mientras que las cepas resistentes restantes se mantuvieron por encima de 83.3%. Resultados que se contrastan con los obtenidos por Guo et al (2019) 41, quienes determinaron la resistencia del bacteriófago vB_PaeM_SCUT- S1 de P. aeruginosa a los pH de 3, 7 y 9; cuyos títulos se mantuvieron, llegando incluso a soportar pH de 11. Según los resultados, los bacteriófagos tienden a mantener una infectividad favorable a pH alcalinos, por ello, es recomendable que, en un eventual uso terapéutico, en su administración, se evite entradas de pH ácidos 42.
Rango de hospedero
Los bacteriófagos reconocen a sus bacterias objetivo con alta especificidad, propiedad mediada por proteínas de unión al receptor (RBP). Estas proteínas son claves en la diversidad taxonómica a la que puede dirigirse un determinado fago, otorgándole su rango de hospederos 43. Los fagos con amplio rango de hospederos, pueden ser potenciales candidatos en fagoterapia. En esta caracterización se evaluó el rango de hospederos de los bacteriófagos aislados frente a 16 cepas de PAMR (Tabla 4). Donde se observó que todas las cepas de bacteriófagos en estudio poseen actividad lítica sobre las cepas PAMR. Se conoce que los bacteriófagos de P. aeruginosa poseen usualmente receptores para un amplio rango de cepas; tal y como se evidencian en el estudio de Santos et al. (2011)11 quienes evaluaron el efecto de los bacteriófagos P5U5 y P2S2 contra 26 cepas de P. aeruginosa aisladas de perros con infecciones oculares oportunistas, donde P5U5 resultó ser activo frente a todas las cepas, mientras que P2S2 lo fue en el 80.8% de ellas (21 cepas). Los rangos de hospederos más amplios obtenidos en este estudio fueron de los bacteriófago F1AO-510 y 506, con un lisado fágico de 9 y 7 (56.25 y 43.75%) de las 16 cepas de PAMR evaluadas (100%); mientras que las cepas restantes, lisaron entre 6 y 4 cepas de PAMR (37.5 y 25%). Cabe resaltar que el lisado parcial (halo turbio) o total (halo claro) de cada cepa fágica, debe ser tomado en cuenta como criterio importante de selección. Los aislados fágicos con amplios rangos de hospederos y en especial, los que producen un lisado total, podrían combinarse para producir "cócteles de fagos" con la gama de hospederos deseado 44.
Los fagos tienden a ser bactericidas en lugar de bacteriostáticos y los que son templados pueden convertirse a menudo en una forma lítica mediante la manipulación genética o la selección natural de mutantes 45, 46. Las infecciones fágicas se realizan durante la fase logarítmica, por ello es importante conocer el tiempo y la concentración de colonias de la bacteria huésped, en esta fase 47. El inicio de la fase exponencial (Log) de la cepa control de P. aeruginosa ATCC 27853 se dio a las cuatro horas de crecimiento, con una densidad óptica (D.O) de 0.096 (Figura 3) y con una concentración de 2.4 x 107 UFC/mL a una longitud de onda de 620 nm en caldo Luria Bertani (LB). Este patrón de crecimiento logarítmico entre la tercera y cuarta hora de P. aeruginosa fue obtenido también por Melvin et al. (2017)48.
Obtención del DNA fágico y análisis RFLPs
Debido a que los fagos son extremadamente diversos en estructura y composición genética, cada aislado debe secuenciarse y caracterizarse adecuadamente antes de que pueda usarse de manera segura en una aplicación médica 49. Para evitar estos problemas, puede ser posible aislar un número limitado de fagos bien caracterizados para unirse e infectar las cepas de interés 50. El método de polimorfismo de longitud de fragmentos de restricción de reacción en cadena de polimerasa (PCR-RFLP) se basa en la digestión de amplicones de PCR con enzimas de restricción apropiadas para producir distintos fragmentos polimórficos utilizados como marcadores para la identificación de especies (51). Muchos estudios han demostrado que la PCR es un método prometedor para la detección de P. aeruginosa, que podría utilizarse en lugar de los métodos convencionales de identificación bioquímica 52. Sin embargo, la variación de la secuencia derivada de la naturaleza altamente polimórfica de su genoma, el intercambio genético con otras especies, así como las diferentes dianas moleculares que no distinguen entre las especies de Pseudomonas estrechamente relacionadas, pueden repercutir en resultados falsos negativos (con los genes ETA, algD oprI y toxA), así como los falsos positivos (con los genes 16S rDNA, 16S-23S rDNA ITS, fliC, oprI y oprL) 53. Por ello, se identificó molecularmente las cepas de PAMR, mediante la técnica de PCR directa cuantitativa, para la detección específica de especie de P. aeruginosa, basada en el gen de la acetilasa del antígeno O (OatA) para un diagnóstico certero 31. La proteína OatA muestra homología con las proteínas de un gran número de especies procariotas y esto define una familia de proteínas de membrana implicadas en la acilación de los restos de carbohidratos exportados. En muchos de estos casos, la acilación define el serotipo o el rango del huésped y por lo tanto, tiene un profundo efecto en la interacción microbio-huésped 54. Así se determinó que las 16 cepas aisladas pertenecen a la especie de P. aeruginosa, tras obtener las bandas de aproximadamente 232 pb, que es el tamaño del amplicón OatA (Figura 10).
La naturaleza del genoma viral del fago F1AO-510, se determinó como DNA, lo cual se demostró al observar la degradación del material genómico al ser tratado con la enzima DNAsa. Al comparar el patrón de restricción producido por la enzima EcoRI, se observó un patrón de bandas diferente tanto en número como en tamaño de las bandas (Figura 11). La restricción del DNA del fago F1AO-510 produjo aproximadamente 11 bandas con un tamaño entre 32 kb, en comparación con el Ladder; mientras que F1AO-513 no produjo bandas, esto nos sugieren que se tratan de dos fagos lítico-diferentes; ya que, en el corrido en gel de electroforesis, el aislados de ADN del fago F1AO-510, fue cortado con la enzima de restricción EcoRI en bandas, representando los productos cortados. mientras que el fago F1AO-513 al no poseer el mismo sitio donde corta la enzima, se observó el DNA no digerido en una sola banda. Estas diferencias en las secuencias de ácidos nucleicos y los sitios donde se unen las enzimas de restricción significan que hay una diferencia en la secuencia entre estas dos cepas. Los RFLPs, son una excelente herramienta para identificar bacteriófagos, permitiendo estimar el peso molecular de su genoma; asimismo posibilita la manipulación del fago desde la tecnología del ADN recombinante. Es recomendable la secuenciación de todo el genoma del bacteriofago aislado F1AO-510, ya que permitiría conocer qué tipo de genes posee, así como el tipo de lisinas que expresa, con miras a un potencial desarrollo de enzibioticos.
Las secuencias de genomas de fagos que no comparten huéspedes raramente comparten similitud de secuencia, aunque esto no es sorprendente en bacteriófagos cuyos huéspedes bacterianos están distantemente relacionados. Por ejemplo, no hay una similitud obvia de secuencias entre genomas de fagos de Streptomyces y mycobacteriófagos, aunque tanto Mycobacterium como Streptomyces son miembros de Actinomycetales. Además, diferentes morfotipos de fagos tienen diferentes organizaciones de sus genomas lo que implica mayor diversidad de secuencias. La gran diversidad genética entre fagos de un huésped común aunado al limitado número de secuencias disponibles es un indicador de la abundancia de secuencias de genomas virales aun no identificados 23. Por lo general, la detección de similitudes de uno o más ORFs predichos con proteínas bien conservadas es suficiente para detectar los módulos, por ejemplo, para detectar el módulo de replicación basta con detectar helicasas y ADN polimerasas 55. El mosaicismo es evidente, aun dentro de las regiones homologas de los genomas de los fagos, es probable que los genes “nuevos” se obtienen de los hospederos y/o de otros grupos de fagos. La extensión del mosaicismo en los genomas del fago es notable y se ha convertido incluso más claro a medida que aumenta el número de genomas disponibles para el análisis comparativo 23. Y una característica importante del mosaicismo es que hace casi imposible rastrear la descendencia exacta de los fagos 55.
CONCLUSIONES
Se lograron aislar y caracterizar macro y microscópicamente 16 cepas de P. aeruginosa multirresistente (PAMR), así como también su identificación genética en el laboratorio, de las muestras de los centros de salud y laboratorios clínicos, asimismo, se determinaron sus perfiles de sensibilidad frente a 13 antimicrobianos, lográndose evidenciar su multirresistencia. Por otro lado, se identificaron ocho lisados fágicos de los aislados PAMR. Se identificó que el nivel de infectividad de los ocho fagos se mantuvo a 4°C por un periodo de 3 meses, disminuyendo a los 6 meses; del mismo modo, se determinó que a -30°C y -80°C, la viabilidad de los fagos mostró una gran variabilidad, observándose que a -80°C, no existe daño sobre ellos, mostrando los fagos FAO-510, FAO-513 y FAO-515, los mejores perfiles de termo estabilidad. Por otro lado, se evidenció que la radiación UV por un periodo de 5 segundos, en algunos fagos no afecto su capacidad infecciosa (F1AO-509 y F1AO-10), sin embargo, a 30 y 60 segundos, si se observó una afección importante en su viabilidad. En su respuesta a altas temperaturas (37°C, 42°C y 47°C) se determinó que, estas afectan su capacidad infectiva, observándose que, a 37°C, hay un descenso marcado en su viabilidad, siendo esta más notoria, conforme incrementa la temperatura, sin embargo, dos fagos (FAO-510 y FAO-514) mostraron tener termo estabilidad, manteniendo su viabilidad. En su capacidad de estabilidad frente a diferentes pH (4, 7 y 9), se pudo ver que a un pH 7, existía una óptima viabilidad, observándose además una tolerancia a pH ácidos de cinco fagos (FAO-502, FAO-509, FAO-510, FAO-513 y FAO-515), siendo el (FAO-513), tolerante a pH alcalino. Se determinó en la prueba de rango de hospedero que los fagos (F1AO-510 y F1AO-506), mostraron una mejor actividad lítica frente a 9 PAMR con 56.25%, y sobre 7 PAMR con 43.75%, siendo los PAMR más susceptibles AO-501 y AO-505, mostrando ser infectados por el 75% de los fagos aislados. Se logró obtener DNA fágico y ser caracterizado molecularmente a través del análisis por RFLPs
Contribución de los autores: «Conceptualización, José Gonzales Cabezas; José Luis Huayán Radas; Ofelia Magdalena Córdova Paz Soldán: Juan Héctor Wilson Krug y Franklin Roger Vargas Vásquez; metodología, José Gonzales Cabezas; José Luis Huayán Radas; Ofelia Magdalena Córdova Paz Soldán: Juan Héctor Wilson Krug y Franklin Roger Vargas Vásquez.; validación, José Gonzales Cabezas; José Luis Huayán Radas; Ofelia Magdalena Córdova Paz Soldán: Juan Héctor Wilson Krug y Franklin Roger Vargas Vásquez.; análisis formal, José Gonzales Cabezas; José Luis Huayán Radas; Ofelia Magdalena Córdova Paz Soldán: Juan Héctor Wilson Krug y Franklin Roger Vargas Vásquez; investigación, José Gonzales Cabezas; José Luis Huayán Radas; Ofelia Magdalena Córdova Paz Soldán: Juan Héctor Wilson Krug; Franklin Roger Vargas Vásquez y Jorge Luis Leiva Piedra; Redacción-redacción del borrador original, José Gonzales Cabezas; José Luis Huayán Radas; Ofelia Magdalena Córdova Paz Soldán: Juan Héctor Wilson Krug y Franklin Roger Vargas Vásquez.; redacción-revisión y edición, José Gonzales Cabezas; José Luis Huayán Radas; Ofelia Magdalena Córdova Paz Soldán: Juan Héctor Wilson Krug y Franklin Roger Vargas Vásquez y Jorge Luis Leiva Piedra.; visualización, José Gonzales Cabezas; José Luis Huayán Radas; Ofelia Magdalena Córdova Paz Soldán: Juan Héctor Wilson Krug y Franklin Roger Vargas Vásquez y Jorge Luis Leiva Piedra. Todos los autores han leído y aceptado la versión publicada del manuscrito»
Conflicto de Intereses: Los autores declaran no tener ningún conflicto de intereses.
REFERENCES
1. Ikpe F, Williams T, Orok E, Ikpe A. Antimicrobial resistance: use of phage therapy in the management of resistant infections. Mol Biol Rep. 21 de agosto de 2024;51(1):925.
2. Bisso-Andrade A. Resistencia bacteriana y el desarrollo de nuevos antimicrobianos. Revista Sociedad Perú de medicina interna. 2012;25(4):2.
3. Das P, Horton R. Physical activity-time to take it seriously and regularly. Lancet. 24 de septiembre de 2016;388(10051):1254-5.
4. Ranjbar R, Alam M. Antimicrobial Resistance Collaborators (2022). Global burden of bacterial antimicrobial resistance in 2019: a systematic analysis. Evid Based Nurs. 27 de julio de 2023;ebnurs-2022-103540.
5. Santamaría Corral G. Terapia con bacteriófagos frente a infecciones causadas por cepas multirresistentes de Pseudomonas aeruginosa [Internet] [http://purl.org/dc/dcmitype/Text]. Universidad Autónoma de Madrid; 2024 [citado 28 de enero de 2025]. p. 1. Disponible en: https://dialnet.unirioja.es/servlet/tesis?codigo=340328
6. Shariati A, Azimi T, Ardebili A, Chirani AS, Bahramian A, Pormohammad A, et al. Insertional inactivation of oprD in carbapenem-resistant Pseudomonas aeruginosa strains isolated from burn patients in Tehran, Iran. New Microbes New Infect. enero de 2018;21:75-80.
7. Remold SK, Brown CK, Farris JE, Hundley TC, Perpich JA, Purdy ME. Differential habitat use and niche partitioning by Pseudomonas species in human homes. Microb Ecol. octubre de 2011;62(3):505-17.
8. Askoura M, Mottawea W, Abujamel T, Taher I. Efflux pump inhibitors (EPIs) as new antimicrobial agents against Pseudomonas aeruginosa. The Libyan journal of medicine. 13 de mayo de 2011;6.
9. Montero MM. Pseudomonas aeruginosa multiresistente: aspectos epidemiológicos, clínicos y terapéuticos [Internet] [http://purl.org/dc/dcmitype/Text]. Universitat Autònoma de Barcelona; 2012 [citado 28 de enero de 2025]. Disponible en: https://dialnet.unirioja.es/servlet/tesis?codigo=76881
10. Breidenstein EBM, de la Fuente-Núñez C, Hancock REW. Pseudomonas aeruginosa: all roads lead to resistance. Trends Microbiol. agosto de 2011;19(8):419-26.
11. Santos T, Ledbetter E, Caixeta L, Bicalho M, Bicalho R. Isolation and characterization of two bacteriophages with strong in vitro antimicrobial activity against Pseudomonas aeruginosa isolated from dogs with ocular infections. American journal of veterinary research. 1 de agosto de 2011;72:1079-86.
12. Venter H. Reversing resistance to counter antimicrobial resistance in the World Health Organisation’s critical priority of most dangerous pathogens. Biosci Rep. 30 de abril de 2019;39(4):BSR20180474.
13. Nicolas-Chanoine MH, Mayer N, Guyot K, Dumont E, Pagès JM. Interplay Between Membrane Permeability and Enzymatic Barrier Leads to Antibiotic-Dependent Resistance in Klebsiella Pneumoniae. Front Microbiol. 2018;9:1422.
14. Poole K. Mechanisms of bacterial biocide and antibiotic resistance. J Appl Microbiol. 2002;92 Suppl:55S-64S.
15. Arzanlou M, Chai WC, Venter H. Intrinsic, adaptive and acquired antimicrobial resistance in Gram-negative bacteria. Essays Biochem. 28 de febrero de 2017;61(1):49-59.
16. Vakulenko SB, Mobashery S. Versatility of aminoglycosides and prospects for their future. Clin Microbiol Rev. julio de 2003;16(3):430-50.
17. Hyman P. Phages for Phage Therapy: Isolation, Characterization, and Host Range Breadth. Pharmaceuticals (Basel). 11 de marzo de 2019;12(1):35.
18. Potera C. Phage renaissance: new hope against antibiotic resistance. Environ Health Perspect. febrero de 2013;121(2):a48-53.
19. Sundar M, G S N, Das DrA, Bhattacharya DrS, Suryan S. Isolation of Host-Specific Bacteriophages from Sewage Against Human Pathogens. Asian Journal of Biotechnology. 1 de abril de 2009;1:163-70.
20. Pimienta E. Tratamiento con bacteriófagos como una alternativa antimicrobiana potencial. Revista CENIC Ciencias Biológicas. 22 de mayo de 2013;44.
21. Gutiérrez D, Martín-Platero AM, Rodríguez A, Martínez-Bueno M, García P, Martínez B. Typing of bacteriophages by randomly amplified polymorphic DNA (RAPD)-PCR to assess genetic diversity. FEMS Microbiol Lett. septiembre de 2011;322(1):90-7.
22. Jo SJ, Kwon J, Kim SG, Lee SJ. The Biotechnological Application of Bacteriophages: What to Do and Where to Go in the Middle of the Post-Antibiotic Era. Microorganisms. septiembre de 2023;11(9):2311.
23. Hatfull GF. Bacteriophage genomics. Current Opinion in Microbiology. 1 de octubre de 2008;11(5):447-53.
24. Reina J, Reina N. [Phage therapy, an alternative to antibiotic therapy?)]. Rev Esp Quimioter. abril de 2018;31(2):101-4.
25. García Salazar IA, Porras Nicho MY. Aislamiento de bacteriófagos de Pseudomonas aeruginosa multidrogo – resistente en aguas de tres ríos de la provincia de Lima-Perú. [citado 28 de enero de 2025]; Disponible en: https://cybertesis.unmsm.edu.pe/item/3a4a6c14-a52e-41d9-9542-a6012f1afc3f
26. Gaviria G, González de S. M, Castaño J. Técnica para aislamiento de bacteriófagos específicos para E.coli DH5á a partir de aguas residuales. Revista MVZ Córdoba. 2012;17(1 (enero-abril)):2852-60.
27. Dini M, Shokoohizadeh L, Jalilian FA, Moradi A, Arabestani MR. Genotyping and characterization of prophage patterns in clinical isolates of Staphylococcus aureus. BMC Res Notes. 21 de octubre de 2019;12(1):669.
28. Mitchell R, Jannasch HW. Processes controlling virus inactivation in seawater. Environ Sci Technol. 1 de octubre de 1969;3(10):941-3.
29. Fortier LC, Moineau S. Phage production and maintenance of stocks, including expected stock lifetimes. Methods Mol Biol. 2009;501:203-19.
30. Wong González A. Purificación y caracterización biológica del bacteriófago uB-19 específico de Bacillus thuringiensis [Internet] [masters]. Universidad Autónoma de Nuevo León; 1994 [citado 28 de enero de 2025]. Disponible en: http://eprints.uanl.mx/325/
31. Choi HJ, Kim MH, Cho MS, Kim BK, Kim JY, Kim C, et al. Improved PCR for identification of Pseudomonas aeruginosa. Appl Microbiol Biotechnol. 1 de abril de 2013;97(8):3643-51.
32. Linzitto O, Arias DO, Cerdá R, Del Curto B, Gatti M, Radman N, et al. Revista de Enfermedades Infecciosas Emergentes (REIE) | Volumen 15. Revista de Enfermedades Infecciosas Emergentes (REIE) [Internet]. 2020 [citado 28 de enero de 2025];15. Disponible en: http://sedici.unlp.edu.ar/handle/10915/132699
33. Ruiz-Garbajosa P, Cantón R. Epidemiology of antibiotic resistance in Pseudomonas aeruginosa. Implications for empiric and definitive therapy. Rev Esp Quimioter. septiembre de 2017;30 Suppl 1:8-12.
34. Hirsch EB, Tam VH. Impact of multidrug-resistant Pseudomonas aeruginosa infection on patient outcomes. Expert Review of Pharmacoeconomics & Outcomes Research. 1 de agosto de 2010;10(4):441-51.
35. Casellas JM. Resistencia a los antibacterianos en América Latina: consecuencias para la infectología. Rev Panam Salud Publica;30(6),dic 2011 [Internet]. 2011 [citado 28 de enero de 2025]; Disponible en: https://iris.paho.org/handle/10665.2/9428
36. Mattila S, Ruotsalainen P, Jalasvuori M. On-Demand Isolation of Bacteriophages Against Drug-Resistant Bacteria for Personalized Phage Therapy. Front Microbiol [Internet]. 13 de noviembre de 2015 [citado 28 de enero de 2025];6. Disponible en: https://www.frontiersin.org/journals/microbiology/articles/10.3389/fmicb.2015.01271/full
37. Luciano P, Jonás R. Caracterización de un bacteriófago específico de Escherichia coli aislado de aguas residuales de “La Taboada”. [citado 28 de enero de 2025]; Disponible en: https://cybertesis.unmsm.edu.pe/item/1ca5cd03-45d4-4bfd-aa0c-c5cf0faeff7a
38. Dirckze CD, Lehrbach PR, Lee BTO. Effect of Bacteriophage C5 on Ultraviolet Light Survival in Pseudomonas aeruginosa. Microbiology. 1979;112(2):297-300.
39. Dominick F, Jesús V de. Aislamiento y caracterización de un bacteriófago con actividad lítica para Vibrio fluvialis. [citado 28 de enero de 2025]; Disponible en: https://cybertesis.unmsm.edu.pe/item/f63168ec-4d14-4815-a9cb-07d4da1fcb84
40. Sonea S. Bacterial Viruses, Prophages, and Plasmids, Reconsidered. Annals of the New York Academy of Sciences. 1987;503(1):251-60.
41. Guo Y, Chen P, Lin Z, Wang T. Characterization of Two Pseudomonas aeruginosa Viruses vB_PaeM_SCUT-S1 and vB_PaeM_SCUT-S2. Viruses. abril de 2019;11(4):318.
42. Honorio-Javes CE, Vallenas-Sánchez Y, Bazán Pérez JT, Honorio-Javes CE, Vallenas-Sánchez Y, Bazán Pérez JT. Bacteriophage cocktail as a substitute for antibiotic-type growth promoters in poultry. Scientia Agropecuaria. octubre de 2021;12(4):499-508.
43. De Jonge PA, Nobrega FL, Brouns SJJ, Dutilh BE. Molecular and Evolutionary Determinants of Bacteriophage Host Range. Trends Microbiol. enero de 2019;27(1):51-63.
44. Gordillo Altamirano FL, Barr JJ. Phage Therapy in the Postantibiotic Era. Clin Microbiol Rev. abril de 2019;32(2):e00066-18.
45. Lenski RE. Experimental evolution and the dynamics of adaptation and genome evolution in microbial populations. ISME J. octubre de 2017;11(10):2181-94.
46. Dedrick RM, Guerrero-Bustamante CA, Garlena RA, Russell DA, Ford K, Harris K, et al. Engineered bacteriophages for treatment of a patient with a disseminated drug-resistant Mycobacterium abscessus. Nat Med. mayo de 2019;25(5):730-3.
47. Fernández L, González S, Campelo AB, Martínez B, Rodríguez A, García P. Low-level predation by lytic phage phiIPLA-RODI promotes biofilm formation and triggers the stringent response in Staphylococcus aureus. Sci Rep. 19 de enero de 2017;7(1):40965.
48. Melvin JA, Gaston JR, Phillips SN, Springer MJ, Marshall CW, Shanks RMQ, et al. Pseudomonas aeruginosa Contact-Dependent Growth Inhibition Plays Dual Role in Host-Pathogen Interactions. mSphere [Internet]. 15 de noviembre de 2017 [citado 29 de enero de 2025]; Disponible en: https://journals.asm.org/doi/10.1128/msphere.00336-17
49. Philipson CW, Voegtly LJ, Lueder MR, Long KA, Rice GK, Frey KG, et al. Characterizing Phage Genomes for Therapeutic Applications. Viruses. abril de 2018;10(4):188.
50. Ando H, Lemire S, Pires DP, Lu TK. Engineering Modular Viral Scaffolds for Targeted Bacterial Population Editing. cels. 23 de septiembre de 2015;1(3):187-96.
51. Oryan A, Yazdi HS, Alidadi S, Doostmohammadi S. Use of a gyrB PCR-RFLP method to diagnose tuberculosis and identify the causative Mycobacterium sp. in cattle and humans. Comparative Immunology, Microbiology and Infectious Diseases. 1 de marzo de 2022;82:101767.
52. Mérault N, Rusniok C, Jarraud S, Gomez-Valero L, Cazalet C, Marin M, et al. Specific Real-Time PCR for Simultaneous Detection and Identification of Legionella pneumophila Serogroup 1 in Water and Clinical Samples. Applied and Environmental Microbiology [Internet]. 1 de marzo de 2011 [citado 29 de enero de 2025]; Disponible en: https://journals.asm.org/doi/10.1128/AEM.02261-10
53. Deschaght P, Van Simaey L, Decat E, Van Mechelen E, Brisse S, Vaneechoutte M. Rapid genotyping of Achromobacter xylosoxidans, Acinetobacter baumannii, Klebsiella pneumoniae, Pseudomonas aeruginosa and Stenotrophomonas maltophilia isolates using melting curve analysis of RAPD-generated DNA fragments (McRAPD). Research in Microbiology. 1 de mayo de 2011;162(4):386-92.
54. Slauch J, Taylor R, Maloy S. Survival in a cruel world: how Vibrio cholerae and Salmonella respond to an unwilling host. Genes Dev. 15 de julio de 1997;11(14):1761-74.
55. Weigel C, Seitz H. Bacteriophage replication modules. FEMS Microbiology Reviews. 1 de mayo de 2006;30(3):321-81.
Received: April 1, 2025 / Accepted: August 6, 2025 / Published: September 15, 2025
Citation: González Cabeza JG, Huayán Radas JL, Córdova Paz Soldán OM, González Jara PS, Vargas Vásquez FR, Leiva Piedra JL. Caracterización molecular y análisis del rango de hospedadores de bacteriófagos líticos dirigidos a Pseudomonas aeruginosa multirresistente aislados de aguas residuales en Perú. Bionatura Journal 2025;2(3):2. doi: 10.70099/BJ/2025.02.03.2
Información adicional. La correspondencia debe dirigirse a jgonzalezc1@upao.edu.pe
Información sobre la revisión por pares. Bionatura agradece a los revisores anónimos su contribución a la revisión por pares de este trabajo utilizando https://reviewerlocator.webofscience.com/.
ISSN.3020-7886
Todos los artículos publicados por Bionatura Journal son de acceso libre y permanente en línea inmediatamente después de su publicación, sin gastos de suscripción ni barreras de registro.
Nota del editor: Bionatura Journal se mantiene neutral en cuanto a las reclamaciones jurisdiccionales en los mapas publicados y las afiliaciones institucionales.
Copyright: © 2025 por los autores. Se presentaron para su posible publicación en acceso abierto bajo los términos y condiciones de la licencia Creative Commons Attribution (CC BY) (https://creativecommons.org/licenses/by/4.0/).




