Resistencia antimicrobiana en bacilos Gram negativos clínicos: caracterización fenotípica y genotípica en Ecuador
Antimicrobial resistance in clinical Gram-negative bacilli: phenotypic and genotypic characterization in Ecuador
Antimicrobial resistance in clinical Gram-negative bacilli: phenotypic and genotypic characterization in Ecuador
RESUMEN
La
resistencia a los antimicrobianos (RAM) es una importante amenaza para la salud
pública mundial, que dificulta el uso de antimicrobianos para el tratamiento de
infecciones bacterianas. En este estudio se caracterizaron los perfiles fenotípicos y genotípicos de
los determinantes de la resistencia a los antimicrobianos en bacterias de
origen clínico procedentes de procesos infecciosos de pacientes atendidos en hospitales de
Quito, Ecuador. Se analizaron 9 aislados bacterianos seleccionados de la Colección Bacteriana -
Quito Católica (CB-QCA) del Laboratorio de Microbiología de la Pontificia
Universidad Católica del Ecuador, preservadas a -20ºC y -80ºC. Se analizaron:
un aislado de E. coli, tres aislados de Klebsiella pneumoniae,
dos Proteus mirabilis y tres Pseudomonas aeruginosa. Las pruebas
de sensibilidad antibiótica fenotípica y las pruebas de mCIM y eCIM se
realizaron mediante el método de difusión en disco. La caracterización
genotípica de los genes de resistencia a betalactámicos y carbapenemasas se
realizó mediante reacción en cadena de la polimerasa (PCR). Los resultados
revelaron una alta prevalencia de resistencia entre los aislados, siendo los
β-lactámicos, las quinolonas y los aminoglucósidos las clases de antibióticos
más comúnmente detectados. Los hallazgos resaltan la necesidad de una
vigilancia continua de los patrones de resistencia a los antimicrobianos.
Keywords:
Enterobacterias; genes de resistencia; betalactámicos;
carbapenemasas; β-lactamasas (BLEE); serin β-lactamasas,
metalo β-lactamasas.
ABSTRACT
Antimicrobial resistance (AMR) is a major global public health threat,
hampering the use of antimicrobials for the treatment of bacterial infections.
In this study, we characterized the phenotypic and genotypic profiles of
antimicrobial resistance determinants in clinically derived bacteria from
infectious processes in patients treated in hospitals in Quito, Ecuador. Nine
bacterial isolates selected from the Catholic Bacterial Collection - Quito
(CB-QCA) of the Microbiology Laboratory of the Pontifical Catholic University
of Ecuador, preserved at -20ºC and -80ºC, were analyzed. One E. coli
isolate, three Klebsiella pneumoniae isolates, two Proteus mirabilis
isolates, and three Pseudomonas aeruginosa isolates were analyzed.
Phenotypic antibiotic susceptibility testing and mCIM and eCIM tests were
performed using the disk diffusion method. Genotypic characterization of
beta-lactam and carbapenemase resistance genes was performed by polymerase
chain reaction (PCR). The results revealed a high prevalence of resistance
among isolates, with β-lactams, quinolones and aminoglycosides being the most
commonly detected antibiotic classes. The findings highlight the need for
continued surveillance of antimicrobial resistance patterns.
Keywords: Enterobacteriaceae; resistance genes; beta-lactams; carbapenemases; β-lactamases (ESBL); serine β-lactamases, metallo β-lactamases.
INTRODUCCIÓN
La
resistencia a los antimicrobianos está aumentando en todo el mundo a niveles
alarmantes y constituye uno de los problemas de
salud más frecuentes en la actualidad.
Según la Organización Mundial de la Salud, OMS, la resistencia antimicrobiana
es una emergencia de salud pública. Actualmente en
todo el mundo se propagan nuevos mecanismos de resistencia bacteriana y ponen
en riesgo la eficacia en el tratamiento de enfermedades infecciosas comunes en
nuestra sociedad1 .
La resistencia a los antimicrobianos (RAM) es la capacidad que tienen
las bacterias, los virus, los hongos y los parásitos de permanecer inmune a los efectos
bactericidas o bacteriostáticos de los antibióticos y otros medicamentos antimicrobianos 1 .
La
resistencia a los antimicrobianos puede ser intrínseca o adquirida. Los mecanismos de resistencia intrínseca son de origen natural y se
deben a los rasgos genéticos propios de cada microorganismo. La resistencia intrínseca es propia
de cada bacteria por ejemplo, todos los Gram negativos son resistentes a la
vancomicina, y esta situación no es variable. Se transmite de generación en
generación de forma vertical. La resistencia adquirida es variable y puede
producirse a través de mutaciones, por cambios en la secuencia de bases del
ADN, o por trasmisión de material genético extracromosómico procedente de otras
bacterias. En este último caso, la transferencia de genes se realiza
horizontalmente a través de plásmidos u otro material genético móvil como
transposones e integrones 2 ,3 .
La transferencia horizontal de genes constituye una ruta altamente
eficaz para la diseminación de genes de resistencia ya que, no solo permite la trasmisión
a otras generaciones, sino también a otras especies bacterianas mediante estas estructuras
genéticas móviles. Donde juega un papel protagónico los plásmidos, transposones
e integrones4 esto que permite la
dispersión de la resistencia y favorece la aparición de brotes a nivel
hospitalario. El uso indiscriminado e inapropiado de antibioticos, combinado
con un control deficiente de infecciones intrahospitalarias, es uno de los
factores que contribuyen a la diseminación de los mecanismos de resistencia5, 6 .
La resistencia
microbiana adquirida ha estado presente en el campo de las enfermedades
infecciosas desde el descubrimiento de la penicilina en 1928 y su producción a
principios de la década de los 40s marcaron los inicios de la era de los
antibióticos en el uso clínico 7 .
Las
enterobacterias son un grupo diverso y ampliamente distribuido en la
naturaleza. El 80% corresponden a los aislamientos Gram negativos en
enfermedades humanas. En general, las
enterobacterias causantes de infecciones han desarrollado mecanismos de
resistencia a los antibióticos betalactámicos. El más importante de estos
mecanismos es la producción de enzimas betalactamasas; estas enzimas son de
naturaleza proteica y son codificadas por genes de origen cromosómico o
plasmidial 5 .
Los antibióticos
betalactámicos representan el principal grupo de antibióticos más seguros,
eficaces y ampliamente utilizados para el tratamiento de infecciones
bacterianas 8 . Estos agentes
actúan inhibiendo la síntesis de la pared celular bacteriana como resultado de
su fuerte unión covalente a las proteínas ligadoras de penicilina PBP (por sus
siglas en inglés “penicillin-binding-proteins”) 9 que catalizan una serie de reacciones de
transpeptidación y carboxilpeptidación necesarias para la síntesis de la pared
celular y además activan una autolisina bacteriana endógena que destruye el
peptidoglicano.10
Los antibióticos
betalactámicos tienen una estructura común llamada anillo betalactámico . La
presencia de grupos sustitutivos unidos al anillo betalactámico establece su
clasificación en: penicilinas, cefalosporinas, carbapenemes y monobactámicos.
Las penicilinas han sido, por largo, tiempo el tratamiento de elección para infecciones humanas. Las cefalosporinas
tienen un gran abanico de acción y pueden ser de primera, segunda, tercera,
cuarta y quinta generación11 .
Los carbapenemes se usan en infecciones intrahospitalarias y en infecciones
causadas por bacterias resistentes a quinolonas, aminoglucósidos y otros
antibióticos de amplio espectro12 .
Se han descrito
alrededor de 8100 betalactamasas13 . Las betalactamasas son enzimas bacterianas que
inactivan a los antibióticos betalactámicos por hidrólisis. Se han clasificado
de acuerdo a dos esquemas generales; la clasificación molecular de Ambler y el
sistema de clasificación funcional de Bush‒Jacoby‒Mideros14 .Para el presente estudio tomaremos en consideración
la clasificación de Ambler.
"Ambler (1980)
propuso una clasificación de betalactamasas en cuatro clases (A, B, C, D) basándose
en similitudes aminoacídicas. Las clases A, C y D son serin-betalactamasas,
mientras que la clase B son metalo-betalactamasas, diferenciándose por el
mecanismo de hidrólisis del anillo betalactámico: serina en el sitio activo
para las primeras y un ion zinc para las últimas 15 .
"Las betalactamasas de clase A, como TEM, SHV,
GES y CTX-M, se encuentran en bacterias Gram-positivas y Gram-negativas,
codificadas en plásmidos y cromosomas." En la clase D, OXA-48 es común en
enterobacterias. Las enzimas de clase C, BLEE y carbapenemasas son de mayor
interes en Enteribcaterias16 .
Las
betalactamasas de clase A son las más diversas, incluyendo enzimas como KPC17 ,
que hidrolizan casi todos los betalactámicos. Las de clase B
(metalo-betalactamasas) como IMP, VIM y NDM son inhibidas por quelantes
metálicos 18 ,19 .
Este estudio tuvo
como objetivo caracterizar los perfiles fenotípicos y genotípicos de los
determinantes de resistencia a antimicrobianos en bacterias clínicas aisladas
de pacientes atendidos en la red hospitalaria de Quito.
Debido a la gran diversidad de estas enzimas que
hidrolizan β-lactámicos y a su
importancia patógena se han planteado los siguientes objetivos en este trabajo.
MATERIALES Y MÉTODOS
Población de estudio y aislados bacterianos
La población de estudio estuvo constituida por nueve aislados bacterianos de origen clínico, previamente seleccionados de la Colección Bacteriana – Quito Católica (CB-QCA) del Laboratorio de Microbiología de la Pontificia Universidad Católica del Ecuador. Se incluyeron: Escherichia coli (n = 1), Proteus mirabilis (n = 2), Klebsiella pneumoniae (n = 3) y Pseudomonas aeruginosa (n = 3). Los aislados seleccionados habían sido caracterizados como productores de β-lactamasas de espectro extendido (BLEE), serin β-lactamasas y metalo-β-lactamasas (MBL).
La población de estudio estuvo constituida por nueve aislados bacterianos de origen clínico, previamente seleccionados de la Colección Bacteriana – Quito Católica (CB-QCA) del Laboratorio de Microbiología de la Pontificia Universidad Católica del Ecuador. Se incluyeron: Escherichia coli (n = 1), Proteus mirabilis (n = 2), Klebsiella pneumoniae (n = 3) y Pseudomonas aeruginosa (n = 3). Los aislados seleccionados habían sido caracterizados como productores de β-lactamasas de espectro extendido (BLEE), serin β-lactamasas y metalo-β-lactamasas (MBL).
Reidentificación de los aislados
Los aislados preservados a −20 °C y −80 °C fueron reactivados en caldo BHI (Brain Heart Infusion, Difco™) e incubados a 37 °C durante 18–24 h. Posteriormente se subcultivaron en agar nutriente, agar eosina azul de metileno (EMB), agar cetramida o agar Hektoen (Difco™) a 37 °C por 24 h.
Los aislados preservados a −20 °C y −80 °C fueron reactivados en caldo BHI (Brain Heart Infusion, Difco™) e incubados a 37 °C durante 18–24 h. Posteriormente se subcultivaron en agar nutriente, agar eosina azul de metileno (EMB), agar cetramida o agar Hektoen (Difco™) a 37 °C por 24 h.
La identificación de enterobacterias se realizó mediante
pruebas bioquímicas convencionales: Citrato de Simmons, Sulfuro-Indol-Motilidad
(SIM), Hierro triple azúcar (TSI), Rojo de metilo (RM), Voges-Proskauer (VP),
Urea, Fenilalanina (FA) y Motilidad-Indol-Lisina (MILI).
En el caso de P. aeruginosa, la identificación
incluyó la prueba de citocromo oxidasa (OxiStrips™, Hardy Diagnostics) y
pruebas complementarias de motilidad, indol y descarboxilación de lisina (MIO,
MILI).
Confirmación por espectrometría de masas
La identificación definitiva de los aislados se realizó mediante espectrometría de masas MALDI-TOF (matrix-assisted laser desorption/ionization, time of flight), reconocida como una herramienta rápida y precisa para la identificación bacteriana20. Los análisis se efectuaron en el Centro de Investigación Microbiológica (Guayaquil, Ecuador).
La identificación definitiva de los aislados se realizó mediante espectrometría de masas MALDI-TOF (matrix-assisted laser desorption/ionization, time of flight), reconocida como una herramienta rápida y precisa para la identificación bacteriana20. Los análisis se efectuaron en el Centro de Investigación Microbiológica (Guayaquil, Ecuador).
Confirmación fenotípica de β-lactamasas
- β-lactamasas
de espectro extendido (BLEE).
La detección de BLEE se realizó mediante el método de difusión en disco, siguiendo las recomendaciones del Clinical and Laboratory Standards Institute (CLSI, 2023)21. Se utilizaron discos de ceftazidima (30 µg) y cefotaxima (30 µg), así como sus combinaciones con ácido clavulánico (30/10 µg). Se interpretó como positivo un incremento ≥5 mm en el halo de inhibición en presencia del inhibidor. - Serin
β-lactamasas (mCIM).
La detección de serin β-lactamasas se llevó a cabo mediante el método Modified Carbapenem Inactivation Method (mCIM). Colonias bacterianas se incubaron en caldo TSB con un disco de meropenem (10 µg) durante 4 h a 35 ± 2 °C. El disco se transfirió luego a placas de MHA inoculadas con E. coli ATCC 25922 (0,5 McFarland). La interpretación de halos de inhibición se realizó de acuerdo con las guías CLSI, 2024. - Metalo
β-lactamasas (eCIM).
Los aislados con sospecha de carbapenemasa fueron confirmados mediante el método EDTA-modified Carbapenem Inactivation Method (eCIM), complementario al mCIM22,23. Se consideró positivo un incremento ≥5 mm en el halo de inhibición en presencia de EDTA.
Confirmación genotípica de β-lactamasas de espectro
extendido (BLEE)
La detección molecular de genes de resistencia se realizó mediante reacción en cadena de la polimerasa (PCR). Los genes analizados incluyeron blaCTX-M, blaTEM y blaSHV24.
La detección molecular de genes de resistencia se realizó mediante reacción en cadena de la polimerasa (PCR). Los genes analizados incluyeron blaCTX-M, blaTEM y blaSHV24.
Las reacciones de PCR (25 µL) incluyeron: 12,5 µL de GoTaq®
Green Master Mix (Promega), 1 µL de cada cebador (forward y reverse), 1 µL de
ADN bacteriano y 9,5 µL de agua grado molecular. Los programas de amplificación
siguieron las especificaciones previamente publicadas. Los productos se
visualizaron en geles de agarosa al 1,5 % teñidos con SYBR®
Gold (Invitrogen).

Tabla 1. Cebadores y condiciones de la
reacción en cadena de la polimerasa para la amplificación de betalactamasas.
Confirmación genotípica de serin β-lactamasas
Las
reacciones de amplificación para los genes codificantes de serin β-lactamasas se emplearon las mismas concentraciones y volúmenes
descritas anteriormente.
Los cebadores empleados en el
proceso de PCR fueron: blaKPC; blaGES; blaOXA-48.
Confirmación genotípica de metalo
β-lactamasas
Las reacciones de amplificación para los genes codificantes de metalo β-lactamasas se emplearon las mismas concentraciones y volúmenes
descritas anteriormente.
Los cebadores
empleados en el proceso de PCR fueron:
blaVIM F, blaVIM R; blaIMP-AF, blaIMP-AR; blaNDM-New-R, blaNDM-New-F.
RESULTADOS
POBLACIÓN
DE ESTUDIO
Se analizaron un total de 9 aislados bacterianos
patógenos procedentes de la Colección Bacteriana – Quito Católica (CB-QCA)
del Laboratorio de Microbiología de la Pontificia Universidad Católica del
Ecuador. La distribución fue la siguiente: Escherichia coli (1/9; 11.1
%), Proteus mirabilis (2/9; 22.2 %), Pseudomonas aeruginosa (3/9;
33.3 %) y Klebsiella pneumoniae (3/9; 33.3 %).
La identificación inicial se realizó mediante pruebas
bioquímicas convencionales y posteriormente fue confirmada por
espectrometría de masas MALDI-TOF, obteniéndose valores de score ≥ 2.0
en todos los casos, lo que garantizó la identificación confiable a nivel de
especie. No se registraron discrepancias entre la identificación bioquímica y
la obtenida por MALDI-TOF.
En cuanto al origen de las muestras clínicas, se
documentó la siguiente distribución: líquido sublingual (1/9; 11.1 %), líquido
peritoneal (1/9; 11.1 %), aspirado traqueal (1/9; 11.1 %), orina (2/9; 22.2 %),
hisopado cloacal de pollo (2/9; 22.2 %), herida (1/9; 11.1 %) y sonda (1/9;
11.1 %).
Confirmación fenotípica de β-lactamasas de espectro extendido
(BLEE) en enterobacterias
Como resultados en los aislados
enterobacterias se encontraron que las 3 Klebsiella pneumoniae
seleccionadas presentaron resistencia Ampicilina (AMP) (penicilina), 2 a la
Amoxicilina/clavulánico (AMC) (agentes combinados de β-lactámicos), 3 a Cefotaxime
(CTX) (cefalosporina) , 2 a Meropenem (MEM), 2 a Imepenem (IMP) (carbapenemes)
, 3 a Aztreonam (ATM) (monobactámico), 3 a Tobramycim (Top) (aminoglucósido) ,
3 Azitromicina (AZM) (nuevo macrólido), y 3 a
Ciprofloxacin (CIP) (Quinolona); aislados intermedios 2 a
Amoxicilina/clavulánico (AMC) (agentes combinados de β-lactámicos), 2 a Imepenem (IMP)
(carbapenemasas) y por ultimo sensibilidad, 1 a meropenem (MEM)
(carbapenemasas) y 3 a Tetraciclina (TE). Con relación a la Escherichia coli se encontraron resistencia:
a la Ampicilina (AMP) (penicilinas), Amoxicilina/clavulánico (AMC) (agentes
combinados de β-lactámicos), Tetraciclina (TE), Cefotaxime (CTX)
(cefalosporina); y muestran sensibilidad al meropenem (MEN), Imepenem (IMP)
(carbapenemasas), Aztreonam (ATM) (monobactámico), Tobramycim (Top)
(aminoglucósido), Azitromicina (AZM) (nuevo macrólido) y Ciprofloxacin (Cip)
(quinolona). De acuerdo a los 2 Proteus mirabilis se registraron resistencia:
2 a la Ampicilina (AMP)(penicilina), 2 a la Tobramycim (Top) (aminoglucósido),
2 a la Tetraciclina (TE), 2 a la Ciprofloxacin (CIP) (quinolona) y 1 a la
Cefotaxime (CTX) (cefalosporina); y sensibilidad a: Amoxicilina/clavulanico (AMC)
(agentes combiandos de β-lactámicos), meropenem (MEM), Imepenem (IMP) (carbapenemasa),
Aztreonam (ATM) (monobactámicos) y Azitromicina (AZM) (nuevo macrólido).
En la identificación de aislados productores de BLEE se realizó con los
antibióticos más el inhibidor acuerdo a las recomendaciones de CLSI:
ceftazidima (30 µg); Ceftazidima/Ácido Clavulánico (30 μg/10 μg); cefotaxima
(30 µg); Cefotaxima/Ácido Clavulánico (30 μg/10 μg); donde se encontró que son
productores de BLEE, presentando un halo de inhibición igual ó superior a 5mm
en relación al antibiótico y al antibiótico y su inhibidor: Ceftazidima y Ceftazidima/Ácido
Clavulánico, así cmo cefotaxima (30 µg) y Cefotaxima/Ácido Clavulánico (30
μg/10 μg). Estos presentaron halos comparativos con su inhibidor superiores a 5
mm.
Confirmación fenotípica de serin β-lactamasas (mCIM)y
metalo β-lactamasas (eCIM)
Mediante la prueba de
mCIM que se detectó serin β-lactamasas se encontró que 5/9 (50,0 %) son
positivos del total de los aislados bacterianos. Donde 2/9 (22,2 %) corresponde
a Klebsiella pneumoniae; 1/9 (11,1 %), P. mirabilis; P.
aeruginosa 2/9 (22,2 %). Y no son productores de serin β-lactamasas 4/9 (40,0
%); 1 aislado de Klebsiella pneumoniae; 1 aislado de E. coli; 1
aislado de P. mirabilis; 1 aislado de P. aeruginosa.
En la prueba de eCIM que se realizó, se identificó que los aislados
bacterianos no son productores de metalo β-lactamasas (Figura 2).
Resistencia bacteriana en Pseudomonas aeruginosa
Se encontró que de los tres aislados de P. aeruginosa analizados
dos son resistentes a todos los antibióticos probados. Se reportó resistencia a
piperacilina/tazobactam (penicilina con agente combinado), Ceftazidime
(cefalosporina), aztreonam (monobactámico), imipenem y meropemen (cabapenemes)
amikacina y tobramicina (aminoglucósido y ciprofloxacina y norfloxacina
(floroquinolonas). Un aislado es sensible a Aztreonam e intermediarimante
resistente a Piperacillin/Tazobactan y Ceftazidime.
Confirmación fenotípica de la producción de β-lactamasas de espectro
extendido (BLEE), serin β-lactamasas (mCIM) y metalo β-lactamasas (eCIM) para los 9 aislados estudiados.
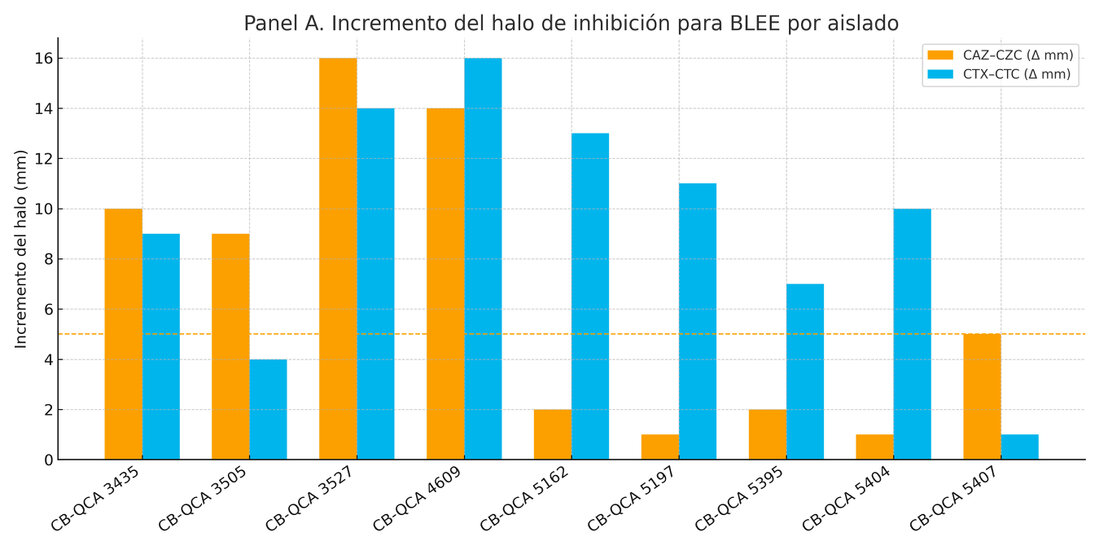
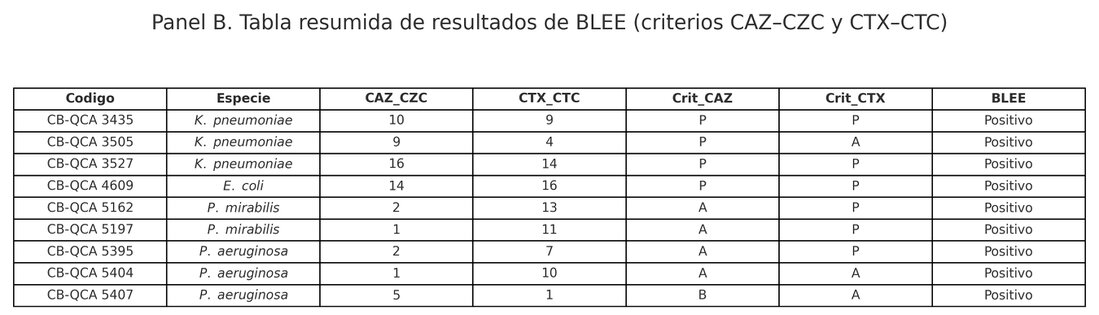
Figura 1. Confirmación
fenotípica de β-lactamasas de espectro extendido (BLEE).
Panel A. Incremento del halo de inhibición (mm) en las pruebas CAZ–CZC y CTX–CTC por aislado bacteriano. La línea discontinua indica el umbral de 5 mm establecido por CLSI para considerar positividad. Los colores representan las diferentes especies bacterianas.
Panel B. Tabla resumen con los valores de incremento de halo y criterios interpretativos (P = positivo, A = ausente, B = borderline). Todos los aislados evaluados fueron positivos para BLEE.
Panel A. Incremento del halo de inhibición (mm) en las pruebas CAZ–CZC y CTX–CTC por aislado bacteriano. La línea discontinua indica el umbral de 5 mm establecido por CLSI para considerar positividad. Los colores representan las diferentes especies bacterianas.
Panel B. Tabla resumen con los valores de incremento de halo y criterios interpretativos (P = positivo, A = ausente, B = borderline). Todos los aislados evaluados fueron positivos para BLEE.

Figura 2. Diagrama de
dispersión del incremento del halo de inhibición (mm) en pruebas de sinergia
CAZ–CZC y CTX–CTC para la detección de BLEE. Cada punto corresponde a un
aislado bacteriano, coloreado según la especie. Las líneas discontinuas marcan
el umbral de ≥5 mm establecido por CLSI como criterio de positividad.

Figura 3.
Detección genotípica de genes de resistencia en aislados bacterianos.
Panel A. Heatmap binario mostrando la presencia (rojo) o ausencia (gris) de genes codificantes de β-lactamasas (blaCTX-M, blaSHV, blaTEM, blaKPC, blaGES, blaOXA, blaVIM, blaIMP y blaNDM) en cada aislado bacteriano (CB-QCA).
Panel B. Frecuencia absoluta de aislados positivos para cada gen analizado.
Panel A. Heatmap binario mostrando la presencia (rojo) o ausencia (gris) de genes codificantes de β-lactamasas (blaCTX-M, blaSHV, blaTEM, blaKPC, blaGES, blaOXA, blaVIM, blaIMP y blaNDM) en cada aislado bacteriano (CB-QCA).
Panel B. Frecuencia absoluta de aislados positivos para cada gen analizado.
Detección molecular de presencia de genes de resistencia en β-lactamasas de espectro extendido (BLEE), serin β-lactamasas y metalo β-lactamasas
La
amplificación de genes mediante PCR, permitió identificar la presencia de genes
productores de BLEE y carbapenemasas. Para la detección de estos genes se
realizó en los geles de agarosa.
De los 9 aislados presentando el gen blaCTX-M el 33,0 %
(3/9); gen blaSHV 56,0 % (5/9); blaTEM
44,0 % (4/9); blaKPC 33,0 % (3/9); blaGES 11,0 % (1/9); blaVIM 33,0 %
(3/9); los blaOXA 0,0 %; blaIMP 0,0 %; blaNDM 0,0 %.
Durante la amplificación de
PCR no se visualizó la presencia de estos genes blaOXA 0,0 %; blaIMP 0.0 %;
blaNDM 0,0 % .
En la Tabla 2 se presentan los
perfiles genotípicos de resistencia detectados en los aislados clínicos de la
colección bacteriana CB-QCA. Se analizaron genes asociados a β-lactamasas de
espectro extendido (blaCTX-M, blaSHV, blaTEM), serin β-lactamasas (blaKPC,
blaGES, blaOXA) y metalo β-lactamasas (blaVIM, blaIMP, blaNDM) (Figura 4).

Tabla 2. Detección
de genes de resistencia β-lactamasa en aislados de la colección bacteriana
CB-QCA (Quito-Católica). P: gen presente; A:
gen ausente.
Para visualizar de manera
comparativa la detección de genes de resistencia, los resultados de PCR
convencional fueron representados mediante un heatmap de presencia/ausencia y
gráficos de barras de frecuencia. Esta aproximación reemplaza la presentación
tradicional de geles de agarosa, permitiendo una interpretación más clara y
cuantitativa de los datos.

Figura 4. Representación
gráfica de genes de resistencia detectados en aislados clínicos.
Panel A: Heatmap de presencia/ausencia de genes β-lactamasa (blaCTX-M, blaSHV, blaTEM, blaKPC, blaGES, blaOXA, blaVIM, blaIMP, blaNDM) en aislados de la colección CB-QCA.
Panel B: Frecuencia de aislados positivos por gen.
Panel C: Distribución de genes de resistencia por aislado (barras apiladas).
Se observa que blaSHV, blaTEM y blaCTX-M fueron los genes más prevalentes, mientras que blaOXA, blaIMP y blaNDM no fueron detectados en los aislados analizados.
Panel A: Heatmap de presencia/ausencia de genes β-lactamasa (blaCTX-M, blaSHV, blaTEM, blaKPC, blaGES, blaOXA, blaVIM, blaIMP, blaNDM) en aislados de la colección CB-QCA.
Panel B: Frecuencia de aislados positivos por gen.
Panel C: Distribución de genes de resistencia por aislado (barras apiladas).
Se observa que blaSHV, blaTEM y blaCTX-M fueron los genes más prevalentes, mientras que blaOXA, blaIMP y blaNDM no fueron detectados en los aislados analizados.
Para confirmar la presencia de genes de
resistencia se realizaron amplificaciones mediante PCR específicas para
diferentes determinantes de β-lactamasas. Los productos amplificados se
analizaron mediante electroforesis en gel de agarosa al 1,5 % teñido con SYBR® Gold (Invitrogen), visualizados bajo
luz UV. A continuación, se muestran geles representativos de algunos genes
detectados en los aislados clínicos estudiados.
Los resultados obtenidos mediante PCR confirmaron la presencia de
genes de β-lactamasas en varios de los aislados estudiados. En particular, blaGES y blaSHV fueron detectados en diferentes proporciones, lo cual concuerda con
lo descrito en otros estudios sobre la diseminación de determinantes genéticos
de resistencia en enterobacterias y Pseudomonas spp. Estos hallazgos refuerzan la importancia del análisis molecular
complementario a las pruebas fenotípicas, permitiendo una caracterización más
precisa de los mecanismos de resistencia presentes en los patógenos clínicos.
DISCUSIÓN
La presente investigación evidencia la preocupante realidad de la
resistencia a antimicrobianos (RAM) en aislados bacterianos clínicos
preservados en el laboratorio de Microbiología de la Pontificia Universidad
Católica del Ecuador (PUCE). Se observó resistencia frente a múltiples familias
de antibióticos de primera línea, incluyendo penicilinas, cefalosporinas,
carbapenémicos, aminoglucósidos, tetraciclinas, macrólidos y fluoroquinolonas.
Este patrón coincide con los reportes internacionales de la OMS y la OPS, que
advierten sobre la diseminación global de la RAM y el riesgo creciente de
infecciones intratables 1,5,6.
En este estudio se incluyeron nueve aislados representativos: Escherichia
coli (11%), Proteus mirabilis (22%), Pseudomonas
aeruginosa (33%) y Klebsiella pneumoniae (33%). Todas
las bacterias fueron identificadas mediante pruebas bioquímicas convencionales
y confirmadas por espectrometría de masas MALDI-TOF, técnica reconocida como
estándar de alta precisión en microbiología clínica 20. Los perfiles
de resistencia observados reflejan lo descrito en Latinoamérica, donde la
prevalencia de enterobacterias productoras de β-lactamasas de espectro
extendido (BLEE) y carbapenemasas se mantiene en aumento 28,29.
Particularmente, se identificaron altos niveles de resistencia en K.
pneumoniae frente a ampicilina, cefotaxima, aztreonam, tobramicina
y ciprofloxacina, lo cual concuerda con reportes de Cuba y Perú, donde cepas
productoras de BLEE superan el 40% en algunos hospitales 27,28. En
Ecuador, el INSPI documentó desde 2010 la emergencia de K.
pneumoniae resistente a carbapenémicos mediada por la enzima KPC,
con una incidencia creciente en los reportes de vigilancia nacional 3,30,31.
Estos hallazgos tienen gran relevancia, pues K. pneumoniae es uno de los
principales reservorios de genes de resistencia transferibles por plásmidos 14–16.
En E. coli, se detectaron resistencias
múltiples pero también sensibilidades residuales frente a carbapenémicos,
hallazgo similar al descrito en la región, donde la prevalencia de BLEE alcanza
cifras de hasta 50% en infecciones urinarias 9,27. P.
mirabilis mostró un perfil intermedio, mientras que P.
aeruginosa presentó los mayores porcentajes de multirresistencia,
con dos aislados resistentes a casi todos los antibióticos probados. Este
comportamiento es consistente con otros estudios que identifican a P. aeruginosa
y Acinetobacter
baumannii como los principales productores de carbapenemasas en
entornos hospitalarios latinoamericanos 17,34.
La
representación mediante heatmap y gráficos facilita la identificación de
patrones de resistencia a nivel genómico. En este estudio, la combinación de
blaCTX-M, blaSHV y blaTEM en varios aislados refuerza la hipótesis de que estos
genes son los principales determinantes de β-lactamasas de espectro extendido
en el contexto latinoamericano 11,24,26–28.
Además, la visualización comparativa permite identificar con claridad los perfiles multigénicos en cepas de K. pneumoniae y P. mirabilis, lo que podría explicar sus fenotipos multirresistentes descritos en la literatura 16,27,29. Este enfoque gráfico constituye una alternativa moderna y más informativa que la presentación de geles de agarosa, y facilita la comparación de resultados entre estudios y centros de vigilancia.
Además, la visualización comparativa permite identificar con claridad los perfiles multigénicos en cepas de K. pneumoniae y P. mirabilis, lo que podría explicar sus fenotipos multirresistentes descritos en la literatura 16,27,29. Este enfoque gráfico constituye una alternativa moderna y más informativa que la presentación de geles de agarosa, y facilita la comparación de resultados entre estudios y centros de vigilancia.
La confirmación genotípica de genes de resistencia reveló la
presencia de blaCTX-M, blaTEM
y blaSHV,
enzimas ampliamente distribuidas en enterobacterias y consideradas los
determinantes más prevalentes de BLEE a nivel mundial 11,24,26.
Estos resultados subrayan la importancia de la transferencia horizontal de
genes mediante plásmidos como mecanismo clave en la rápida diseminación de la
resistencia 14,15. Sin embargo, no se detectaron genes blaOXA,
blaIMP
ni blaNDM,
lo que puede atribuirse a variabilidad genética, mutaciones en sitios de unión
de los cebadores o baja expresión génica, factores descritos previamente como
limitaciones de la PCR 35–38.
Los resultados de este estudio se enmarcan en una problemática de
gran impacto en salud pública. La RAM se proyecta como una de las principales
causas de mortalidad global hacia el año 2050, con cifras estimadas de hasta 10
millones de muertes anuales si no se implementan medidas efectivas 1,32.
En España y Europa, se calcula que las infecciones por bacterias
multirresistentes ocasionan más de 33 000 muertes al año, con un elevado costo
sanitario 32,33.
En el contexto ecuatoriano, la vigilancia nacional ha mostrado que E. coli
es el principal microorganismo resistente (61%), seguido de K.
pneumoniae, Staphylococcus aureus y P.
aeruginosa 30,31. Esta tendencia coincide con lo
observado en este trabajo, donde E. coli y K.
pneumoniae fueron los aislados más representativos.
En conjunto, los hallazgos de esta investigación refuerzan la
necesidad de fortalecer los programas de vigilancia microbiológica en Ecuador,
promover el uso racional de antibióticos y articular esfuerzos bajo el enfoque One
Health, integrando salud humana, animal y ambiental 5,32.
La implementación de estrategias como los programas de administración de
antimicrobianos (antimicrobial stewardship), la
mejora de las prácticas de higiene hospitalaria y el desarrollo de nuevas
moléculas antibacterianas resultan fundamentales para contener esta amenaza 6,16,32.
Limitaciones
Este estudio presenta algunas limitaciones. En primer lugar, el
tamaño de la muestra fue reducido (n = 9 aislados), lo que limita la
posibilidad de extrapolar los hallazgos a la población bacteriana circulante en
otras regiones del país. En segundo lugar, si bien la PCR permitió identificar
genes de resistencia clave, la ausencia de detección de genes como blaOXA,
blaIMP
y blaNDM
podría deberse a mutaciones en las secuencias diana, baja expresión génica o
variabilidad regional, lo que puede generar falsos negativos 35–38.
Finalmente, no se evaluaron variables clínicas asociadas, como la respuesta
terapéutica de los pacientes o los desenlaces clínicos, lo cual restringe la
correlación de los hallazgos microbiológicos con el impacto clínico real.
Perspectivas
Futuros estudios deberían incluir un mayor número de aislados y
abarcar diferentes centros de salud del país para obtener un panorama más
representativo. Además, se recomienda el empleo de técnicas de secuenciación de
nueva generación (NGS) y paneles multiplexados, que ofrecen una mejor
resolución en la identificación de variantes genéticas y permiten superar las
limitaciones de la PCR convencional 35–37. Integrar la
caracterización microbiológica con información clínica de los pacientes
fortalecerá la aplicabilidad de los resultados en el manejo terapéutico y en
las políticas de control de infecciones.
A pesar de estas limitaciones, este trabajo constituye un aporte
novedoso en la vigilancia de la RAM en Ecuador, al combinar pruebas
fenotípicas, genotípicas y confirmación por MALDI-TOF. Los hallazgos obtenidos
proporcionan una base sólida para diseñar estudios multicéntricos de mayor
escala y para apoyar los programas nacionales de vigilancia y control de
resistencia antimicrobiana en concordancia con el enfoque One
Health.
CONCLUSIONES
Los resultados de
esta investigación confirman la elevada prevalencia de resistencia a
antimicrobianos (RAM) en bacterias patógenas de importancia clínica. La
resistencia se observó principalmente frente a antibióticos de las familias de
los β-lactámicos, quinolonas y aminoglucósidos, lo que representa una amenaza
significativa para el tratamiento de infecciones comunes y refuerza la
necesidad de una vigilancia continua en el contexto local y regional.
El análisis de
los nueve aislados clínicos seleccionados permitió caracterizar la distribución
de especies con un predominio de Klebsiella pneumoniae y Pseudomonas
aeruginosa, seguidas de Proteus mirabilis y Escherichia coli.
Todas las bacterias fueron identificadas mediante pruebas bioquímicas
convencionales y confirmadas por espectrometría de masas MALDI-TOF, lo que
asegura la precisión de la identificación y constituye un aporte metodológico
relevante en la caracterización de la RAM en Ecuador.
Un aspecto
novedoso de este trabajo es el enfoque integral empleado para la
caracterización de los aislados. A nivel fenotípico, se corroboró la producción
de β-lactamasas de espectro extendido (BLEE), serin β-lactamasas y metalo
β-lactamasas, confirmando la capacidad de los patógenos para inactivar
diferentes familias de antibióticos. A nivel genotípico, la detección de genes
de resistencia como blaCTX-M, blaTEM y blaSHV mediante PCR
aporta una comprensión más profunda de los mecanismos moleculares que sustentan
la resistencia en los aislados estudiados.
En conjunto, este
estudio constituye uno de los primeros esfuerzos en Ecuador en integrar métodos
fenotípicos, genotípicos y de espectrometría de masas para la caracterización
de bacterias multirresistentes. Esta aproximación multidimensional no solo consolida
los objetivos planteados, sino que también ofrece una base sólida para futuras
investigaciones multicéntricas y para el fortalecimiento de los programas de
vigilancia de la resistencia antimicrobiana en el país bajo un enfoque One
Health.
REFERENCIAS
1.
Organización
Mundial de la Salud (OMS). La OMS publica un informe sobre el estado de
desarrollo de antibacterianos [Internet]. OMS; 2024 [cited 2024 Sep 3].
Available from: https://www.who.int/es/news/item/14-06-2024-who-releases-report-on-state-of-development-of-antibacterials
2.
Camacho-Silvas
LA. Resistencia bacteriana, una crisis actual. Rev Esp Salud Publica.
2023;97:e202311123.
3.
Instituto
Nacional de Investigación en Salud Pública (INSPI). Reporte de datos de
resistencia a los antimicrobianos en Ecuador 2014–2018. Ministerio de Salud
Pública; 2018. p. 1–10.
4.
da
Silva JD, Bens L, Santos AJ do C, Lavigne R, Soares J, Melo LDR, et al.
Isolation and characterization of the Acadevirus members BigMira and MidiMira
infecting a highly pathogenic Proteus mirabilis strain. Microorganisms.
2023;11(9):2141.
5.
Organización
Mundial de la Salud. Semana Mundial de Concientización sobre el Uso de los
Antimicrobianos 2021 [Internet]. OMS; 2021 [cited 2024 Sep 3]. Available from: https://www.paho.org/es/campanas/semana-mundial-concientizacion-sobre-uso-antimicrobianos-2021
6.
Organización
Panamericana de la Salud (OPS). Un informe pone de relieve el aumento de la
resistencia a los antibióticos en infecciones bacterianas que afectan al ser
humano. OPS; 2022 Dec 9 [cited 2024 Sep 3]. Available from: https://www.paho.org/es/noticias/9-12-2022-informe-pone-relieve-aumento-resistencia-antibioticos-infecciones-bacterianas
7.
Espinoza
González AG, Pineda Sánchez DE. 95 años del descubrimiento de la penicilina.
Rev Med Hondur. 2023;91(Supl.2):54–8.
8.
Zurita
J. Resistencia bacteriana en el Ecuador. Rev Panam Infectol. 2012;14(2):75–81.
9.
Sosa
Flores JL, Chapoñan Mendoza JF. Resistencia antibiótica de Escherichia coli
según producción de betalactamasas de espectro extendido en urocultivos,
Hospital III-1 Chiclayo, Perú 2020. Rev Cuerpo Med HNAL Almanzor Aguinaga
Asenjo. 2023;15(4):598–603.
10.
Shlaes
DM. Antibiotics: The perfect storm. Springer; 2010.
11.
Bonnet
R. Growing group of extended-spectrum β-lactamases: the CTX-M enzymes.
Antimicrob Agents Chemother. 2004;48(1):1–14.
12.
Otoya
Durand E, García-Mostajo JA, Rivera-Morón P. Tendencia en el consumo de
carbapenems en un hospital de asociación público-privada. Rev Cuerpo Med HNAL
Almanzor Aguinaga Asenjo. 2021;14(2):92–8.
13.
Anderson
R, Groundwater P, Todd A, Worsley A. Antibacterial agents: chemistry, mode of
action, mechanisms of resistance and clinical applications [Internet]. Wiley;
2012 [cited 2024 Sep 3]. Available from: https://www.wiley.com/en-us/Antibacterial+Agents%3A+Chemistry%2C+Mode+of+Action%2C+Mechanisms+of+Resistance+and+Clinical+Applications-p-9780470748214
14.
Ambler
RP, Coulson AFW, Frère JM, Ghuysen JM, Joris B, Forsman M, et al. A standard
numbering scheme for the class A β-lactamases. Biochem J. 1991;276(1):269–70.
15.
Astocondor-Salazar
L. Betalactamasas: la evolución del problema. Rev Peru Investig Salud.
2018;2(2):42–9.
16.
Lepe
JA, Martínez-Martínez L. Mecanismos de resistencia en bacterias gramnegativas.
Med Intensiva. 2022;46(7):392–402.
17.
Pintos-Pascual
I, Cantero-Caballero M, Rubio EM, Sánchez-Romero I, Asensio-Vegas Á,
Ramos-Martínez A. Epidemiología y clínica de las infecciones y colonizaciones
por enterobacterias productoras de carbapenemasas en un hospital de tercer
nivel. Rev Esp Quimioter. 2020;33(2):123–30.
18.
Clinical
and Laboratory Standards Institute (CLSI). Performance standards for
antimicrobial susceptibility testing. 33rd ed. CLSI supplement M100. Wayne, PA:
CLSI; 2023.
19.
Lasko
MJ, Gill CM, Asempa TE, Nicolau DP. EDTA-modified carbapenem inactivation
method (eCIM) for detecting IMP metallo-β-lactamase-producing Pseudomonas
aeruginosa: an assessment of increasing EDTA concentrations. BMC Microbiol.
2020;20(1):308.
20.
Di
Conza JA. Aplicaciones de la espectrometría de masas MALDI-TOF en la
microbiología clínica. Rev Argent Microbiol. 2022;54(3):163–5.
21.
Clinical
and Laboratory Standards Institute (CLSI). Performance standards for
antimicrobial susceptibility testing. 34th ed. CLSI supplement M100. Wayne, PA:
CLSI; 2024.
22.
Tsai
YM, Wang S, Chiu HC, Kao CY, Wen LL. Combination of modified carbapenem
inactivation method (mCIM) and EDTA-CIM (eCIM) for phenotypic detection of
carbapenemase-producing Enterobacteriaceae. BMC Microbiol. 2020;20(1):315.
23.
Koul
N, Kakati B, Agarwal S. Use of the combined modified carbapenem inactivation
method and EDTA-modified carbapenem inactivation method for detection of
carbapenemase-producing Enterobacteriaceae causing ventilator-associated
respiratory infections. J Pure Appl Microbiol. 2022;16(2):1239–44.
24.
El
Aila NA, Al Laham NA, Ayesh BM. Prevalence of extended spectrum β-lactamase and
molecular detection of blaTEM, blaSHV and blaCTX-M genotypes among
Gram-negative bacilli isolates from pediatric patients in Gaza Strip. BMC
Infect Dis. 2023;23(1):99.
25.
Kibbe
WA. OligoCalc: an online oligonucleotide properties calculator. Nucleic Acids
Res. 2007;35(Web Server):W43–6.
26.
Yauri
Bucheli MF, Alcocer I, Duncan C. Caracterización molecular de genes de
resistencia a β-lactámicos en aislados bacterianos clínicos de la familia
Enterobacteriaceae. Rev Ecuat Med Cienc Biol. 2021;42(1):45–54.
27.
Gonzales-Rodriguez
AO, Infante Varilla SF, Reyes-Farias CI, Ladines Fajardo CE, Gonzales Escalante
E. β-lactamasas de espectro extendido y factores de virulencia en Escherichia
coli uropatógenas en asilos de ancianos en Lima, Perú. Rev Peru Med Exp Salud
Publica. 2022;39(1):98–103.
28.
Valdez
Fernandez-Baca LM. Escherichia coli productoras de β-lactamasas de espectro
extendido (BLEE), un problema creciente en nuestros pacientes. Rev Med Hered.
2017;28(3):180–6.
29.
Monté
Cepero L, Martínez CR. Escherichia coli y Klebsiella pneumoniae productoras de
β-lactamasas de espectro extendido en un hospital de La Habana. Rev Cubana Hig
Epidemiol. 2022;60(2):e1754.
30.
Instituto
Nacional de Investigación en Salud Pública (INSPI). Reporte de datos de
resistencia a los antimicrobianos en Ecuador 2014–2018 [Internet]. Ministerio
de Salud Pública; 2019 [cited 2024 Sep 9]. Available from: https://www.salud.gob.ec/wp-content/uploads/2019/08/gaceta_ram2018.pdf
31.
Mendieta
Astudillo V, Gallegos Merchan JD, Peña Cordero SJ. Frecuencia de BLEE, AmpC y
carbapenemasas en urocultivos de Escherichia coli de origen comunitario. Rev
Vive. 2021;4(11):387–96.
32.
Ministerio
de Sanidad, España. One Health High-Level Meeting on Antimicrobial Resistance.
Plan Nacional frente a la Resistencia a los Antibióticos (PRAN). 2023.
33.
Agencia
Española de Medicamentos y Productos Sanitarios (AEMPS). Productos sanitarios
para diagnóstico in vitro. Informe técnico COVID-19. 2022.
34.
Quiroz
R, Sánchez A, Ramos L, Delgado C. Resistencia a carbapenémicos y producción de
carbapenemasas en bacilos Gram negativos aislados en el hospital Belén de
Trujillo, Perú (2018–2019). Rev Cuerpo Med HNAL Almanzor Aguinaga Asenjo.
2020;14(3):201–9.
35.
Opazo
A, et al. Diagnóstico molecular para la detección genotípica de la resistencia
a los antibióticos: Panorama actual y direcciones futuras. J Antimicrob
Chemother. 2023;78(5):1123–35.
36.
Antibiotics
(Basel). Tecnologías actuales y futuras para la detección de bacterias
resistentes a los antibióticos. Antibiotics (Basel). 2023;12(4):456.
37.
Journal
of Clinical Microbiology. Detección rápida de Enterobacterales productoras de
carbapenemasas: una revisión sistemática de los métodos de diagnóstico y su
impacto clínico. J Clin Microbiol. 2021;59(12):e02534–21.
38.
Zhang
Y, Chen H, Li Q. Structure-based primer design minimizes PCR failures caused by
SARS-CoV-2 mutations. Pathogens. 2021;10(6):748.
Received: May 14, 2025 / Accepted: August 20, 2025 / Published:
September 15, 2025
Citation: Tamami Tualombo SB, Yauri Bucheli MF, Alcocer Negrete IR.
Resistencia antimicrobiana en bacilos Gram negativos clínicos: caracterización
fenotípica y genotípica en Ecuador. Bionatura Journal. 2025;2(3):13.
doi:10.70099/BJ/2025.02.03.13
Additional
Information
Correspondence should be addressed to: sandrytatu@gmail.com
Correspondence should be addressed to: sandrytatu@gmail.com
Peer
Review Information. Bionatura Journal thanks the
anonymous reviewers for their contribution to the peer review of this work
using Reviewer Locator.
ISSN: 3020-7886
All
articles published in Bionatura Journal are freely and
permanently accessible online immediately after publication, with no
subscription charges or registration barriers.
Editor’s
Note: Bionatura Journal remains neutral
with regard to jurisdictional claims in published maps and institutional
affiliations.
Copyright: © 2025 by the authors. Submitted for possible open access
publication under the terms and conditions of the Creative Commons Attribution
(CC BY) license (https://creativecommons.org/licenses/by/4.0/).




