De la microscopía a la secuenciación genética: La evolución en las técnicas de diagnóstico de la Leucemia Linfoide Aguda
From Microscopy to Genetic Sequencing: The Evolution of Diagnostic Techniques for Acute Lymphoblastic Leukemia
Dayana Fernanda Pico Sánchez 1, Daniela Alexandra Rosero Freire 2*
1 Universidad Técnica de Ambato, Facultad de Ciencias de la Salud, Carrera de Laboratorio Clínico. Ambato, Ecuador; dpico4968@uta.edu.ec.
2 Universidad Técnica de Ambato, Facultad de Ciencias de la Salud, Carrera de Laboratorio Clínico. Ambato, Ecuador
* Correspondence: da.roserof@uta.edu.ec
RESUMEN
La Leucemia Linfoide Aguda (LLA) es una enfermedad
hematológica muy heterogénea que afecta tanto a niños como adultos, cuyas tasas
de curación han incrementado con el pasar de los años. Esto se debe a la
evolución en las técnicas de diagnóstico, que han contribuido a realizar una
detección más temprana y precisa, monitorear la evolución de la LLA y reconocer
el pronóstico. El objetivo del presente artículo es proporcionar una revisión
comprensiva y actualizada sobre los avances en las técnicas utilizadas para el
diagnóstico de la LLA. Se encontró que el diagnóstico parte de los resultados
dados por las técnicas convencionales como el hemograma y el análisis de la
morfología celular. Sin embargo, estas deben ser complementadas por técnicas
más avanzadas como el cariotipo, Fluorescence in Situ Hybridization (FISH),
RT-PCR que detectan alteraciones y mutaciones a nivel molecular. Además de
otras técnicas como la NGS, que, aunque aún se limitan a los laboratorios de
investigación siguen brindando información útil. Estos avances han mejorado
significativamente la identificación de subtipos moleculares y alteraciones
genéticas, las cuales son clave para la estratificación de riesgo y el
pronóstico, aunque no se puede dejar atrás a las técnicas convencionales, ya que
son el punto de partida para la realización de análisis posteriores.
Palabras Clave: cariotipo convencional; FISH; hemograma; Leucemia
Linfoide Aguda; NGS; RT-PCR.
ABSTRACT
Acute Lymphoblastic Leukemia (ALL)
is a highly heterogeneous hematological disease that affects both children and
adults, with cure rates that have increased over the years. This progress is
attributed to advancements in diagnostic techniques, which have contributed to
earlier and more precise detection, monitoring of ALL progression, and
prognosis assessment. The objective of this article is to provide a
comprehensive and updated review of the advances in techniques used for
diagnosing ALL. It was found that diagnosis begins with results obtained from
conventional techniques, such as complete blood count and cellular morphology
analysis. However, these must be complemented by more advanced techniques, such
as karyotyping, FISH, and RT-PCR, which detect molecular-level alterations and
mutations. Additionally, other techniques like NGS, although still limited to
research laboratories, continue to provide valuable information. These
advancements have significantly improved the identification of molecular subtypes
and genetic alterations, which are crucial for risk stratification and
prognosis. Nonetheless, conventional techniques remain indispensable as the
starting point for subsequent analyses.
Keywords: conventional karyotyping; FISH; complete blood
count; Acute Lymphoblastic Leukemia; NGS; RT-PCR.
INTRODUCCIÓN
La leucemia linfocítica aguda (LLA) es una enfermedad
hematológica muy heterogénea que abarca una variedad de neoplasias linfoides
que se desarrollan a partir de células precursoras de los linajes B y T 1 . Estas neoplasias pueden presentarse
principalmente como un proceso leucémico generalizado que afecta tanto la médula
ósea (MO) como la sangre periférica, o pueden aparecer con infiltración
localizada en los tejidos, con una afectación mínima de la MO 2 .
Entre los factores de riesgo de esta enfermedad, se
pueden mencionar la exposición a radiación antes y después del nacimiento,
mutaciones genéticas como el síndrome de Down, tratamientos con quimioterapia,
infecciones virales como el virus linfotrópico humano de células T tipo 1
(HTLV1), el virus de Epstein-Barr (EBV), o tener un gemelo idéntico con LLA,
entre otros 3 . También este tipo de leucemia tiene su
pico etario entre los 2 y 5 años 4 , representando el cáncer más frecuente en
pacientes pediátricos, siendo mucho menos común en adolescentes, adultos
jóvenes y adultos mayores 5 .
A nivel mundial los casos de LLA aumentaron de 49,1
mil en 1990 a 64,2 mil en el año 2017, mientras que la tasa de incidencia
ajustada por edad disminuyó levemente. Es importante mencionar también que la
mayor frecuencia de diagnóstico en niños y adultos jóvenes fue registrada en
regiones en vías de desarrollo. Un ejemplo de ello es que >20% de los casos
totales de LLA se han reportado en regiones de bajo índice sociodemográfico,
mientras que en América Latina central representaron >30% 6 .
En los últimos años la incidencia de la LLA de células
B (LLA-B) ha aumentado a nivel global en comparación con el linaje de células T
(LLA-T), especialmente en América Latina, debido a diversos factores como
ambientes laborales que no cumplen con los estándares de control a exposiciones
cancerígenas, la migración a lugares con peores condiciones ambientales, e
incluso la exposición a nuevos agentes infecciosos 4 .
En estos países las tasas de curación varían
ligeramente, por ejemplo, en Chile y Argentina los porcentajes de supervivencia
son de más del 70%, mientras que en México se estima que son de alrededor del
50% 7 . En contraparte, las tasas de curación en
países desarrollados superan el 90% en la mayoría de los casos infantiles y del
30 al 40% en el caso de pacientes adultos, esta significativa mejora se ha dado
debido a los avances en el diagnóstico clínico y en la comprensión de la LLA 5 .
Los métodos tradicionales de diagnóstico de la LLA han
sido fundamentales tanto para la detección como para el tratamiento de la
enfermedad. Desde un principio el diagnóstico de la LLA se iniciaba con el
hemograma, donde se analizaban parámetros como la hemoglobina (Hb), hematocrito
(Hct), recuento de glóbulos rojos (RBC), índices hematimétricos, contaje total
de leucocitos (WBC) y plaquetas (PCT) 8 , además de los análisis microscópicos que
examinaban la morfología celular en busca de blastos y las tinciones
citoquímicas, utilizadas principalmente para poder diferenciar la LLA de la
leucemia mieloide aguda (LMA) 5,9 . No obstante, en la actualidad, estas
características morfológicas rara vez son útiles para diferenciar los subtipos
de la enfermedad, ya que, aunque puede detectar células leucémicas no
proporcionan suficiente información para que los médicos confirmen el
diagnóstico de esta enfermedad 10,11 .
Debido a estas limitaciones, se emplean otros métodos
como la inmunofenotipificación a través de citometría de flujo o la
inmunohistoquímica, cuyo objetivo es confirmar que las células leucémicas sean
de origen linfoide 5 . Asimismo, la necesidad de obtener una
comprensión más amplia de la enfermedad ha impulsado el desarrollo y la
aplicación de métodos enfocados en la genética, los cuales han permitido la
identificación de nuevos subtipos moleculares de la LLA y el descubrimiento de
nuevas alteraciones o mutaciones genéticas que predisponen al desarrollo de
esta enfermedad 12 .
El presente artículo tiene como objetivo proporcionar
una revisión exhaustiva de la evolución que han tenido las técnicas de
diagnóstico de la LLA. A través de un análisis detallado de la literatura, se
busca resaltar las limitaciones de las técnicas convencionales, como el
hemograma y el análisis de frotis, así como el potencial de otras metodologías,
incluyendo la citometría de flujo, la inmunohistoquímica y los nuevos enfoques
basados en la genética. Este trabajo pretende no solo ofrecer una visión integral
de la evolución del diagnóstico de la LLA, sino también subrayar la importancia
de una detección temprana y precisa para mejorar los resultados clínicos en los
pacientes.
Técnicas convencionales o tradicionales en diagnóstico
de LLA
Hemograma
En la gran mayoría de casos, el diagnóstico de la LLA
gira en torno al hemograma completo (CBC, por sus siglas en inglés), debido a
las situaciones clínicas que derivan a solicitar este análisis, además de los
datos relevantes que se pueden obtener del mismo 13 . De acuerdo con los parámetros analizados
en el CBC la LLA cursa con anemia normocítica normocrómica; trombocitopenia,
aunque uno de cada cinco niños no presenta esta alteración. En cuanto a los
glóbulos blancos estos pueden encontrarse con niveles altos, bajos o normales,
siendo así que, el 50% de los niños presentan contajes de menos de 11.000
leucocitos/μl en el hemograma inicial, el 20% tienen
más de 100.000/μl y solo el 7% presenta contajes de 200.000 células/μl 3,13 . En cuanto a los adultos, los contajes de
leucocitos rondan entre 30.000 a 100.000 células/μl 14 . En
el frotis de sangre periférica el 90% de los pacientes suelen presentar células
blásticas 15 .
Análisis morfológico en médula ósea o sangre
periférica
En el estudio de la MO es necesario demostrar la
existencia de más del 25% de linfoblastos, además la médula tiene que presentar
entre otras características, la hipercelularidad con una notable sustitución de
los espacios adiposos y otros componentes medulares normales por células
leucémicas, también se pueden observar algunos precursores mieloides y
eritroides aparentemente normales y megacariocitos normales o ausentes9 .
Por otra parte, el análisis de la morfología de las
células sanguíneas permaneció como la principal herramienta empleada en el
abordaje de la leucemia aguda para distinguir si esta era de origen
linfoblástico o mieloblástico16 . La clasificación más aceptada respecto a
la morfología celular es la planteada por la asociación FAB
(Franco-América-Británica), la cual analiza las características de las células
luego de ser coloreadas por la tinción de Wright17 o la tinción May-Grunwald-Giemsa15 . Con estos parámetros la LLA es
clasificada en 3 grupos: L1, L2 y L3, de acuerdo con el diámetro celular, la
forma del núcleo, la cantidad y prominencia de los nucléolos, así como la
proporción y aspecto del citoplasma9 .

Tabla 1. Características morfológicas de los linfocitos en la LLA.9,15
Tinciones Citoquímicas
Utilizadas para poder diferenciar la LLA de la
leucemia mieloide aguda (LMA), las principales reacciones que van a servir para
realizar esta diferenciación son la mieloperoxidasa y el sudan negro18 , ya que los linfoblastos son completamente
negativos9 . Los linfoblastos T muestran actividad
paranuclear en la esterasa inespecífica cuando se realiza en un pH ácido, con
más del 75% de actividad en la fosfatasa ácida. En la tinción de ácido
periódico de Schiff (PAS), los linfoblastos generalmente presentan una
coloración distintiva con gránulos gruesos o bloques masivos en forma de
anillos concéntricos. Además, es más común encontrar una reacción PAS negativa
en la LLA de linaje T que en la de linaje B9 .
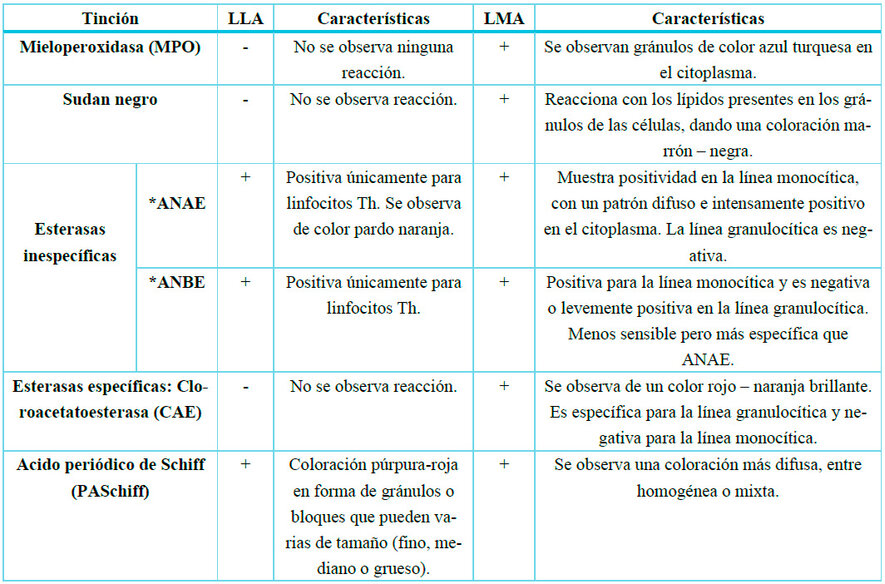
*ANAE: α-naftil acetato esterasa; *ANBE: α-naftil butirato esterasa.
Tabla 2. Características diferenciales
microscópicas de las tinciones citoquímicas 19 .
Inmunofenotipificación
Actualmente, de acuerdo con la Organización Mundial de
la Salud (OMS) las leucemias agudas se clasifican a partir del linaje, ya sea
mieloide o linfoide, lo cual se determina mediante la presencia de ciertos
antígenos en las células malignas20 . La inmunofenotipificación representa uno
de los métodos más importantes para clasificar correctamente a la LLA y para
ello se utilizan dos técnicas diferentes: la inmunohistoquímica (IHQ) para el
estudio de la biopsia de MO y la citometría de flujo (CF) en el análisis del
aspirado medular21 .
Inmunohistoquímica
La IHQ detecta la interacción entre el antígeno y el
anticuerpo mediante un cambio colorimétrico, que puede observarse a través de
un microscopio óptico o bien con marcadores de fluorescencia utilizando un
microscopio con este mismo enfoque. Esta técnica se utiliza en secciones de
tejidos fijados con formaldehído e incrustados en parafina, y habitualmente se
emplea un único anticuerpo por sección15,22 . En un principio, se vio limitada por el
uso de anticuerpos de baja afinidad o interpretaciones erróneas, sin embargo,
actualmente y con los avances tecnológicos realizados, la IHQ es mucho más útil
en el análisis de células que son demasiado frágiles como para soportar el
procesamiento de la muestra usando la CF. Entre estos avances se destacan la
recuperación de antígenos, implementación de equipos automatizados de tinción y
la producción comercial de anticuerpos20 . Basándose en estos puntos, la IHQ se ha
convertido en una técnica mucho más accesible y que proporciona resultados
mucho más rápidos manteniendo e incluso mejorando la precisión de estos.
El primer paso en este proceso es la recuperación del
antígeno, lo que implica un pretratamiento del tejido para recuperar los
antígenos ocultos por el proceso de fijación y así facilitar su acceso para la
unión con los anticuerpos. Posteriormente, se añade el anticuerpo primario que
se va a unir al antígeno de interés, se lava el exceso de anticuerpos no unidos
y se aplica el antígeno secundario conjugado con enzimas (como la peroxidasa de
rábano picante) o con marcadores fluorescentes. En el caso de que se trate con
una enzima es necesario añadir un cromógeno (por ejemplo, NovaRED) para
visualizar el cambio de color en un microscopio óptico. Finalmente, se realiza
una contratinción con hematoxilina seguido de un proceso de deshidratación,
aclarado y montaje para el análisis de la muestra, ya sea en un microscopio
óptico o de fluorescencia22 .

Figura 1. Fundamento de la inmunohistoquímica basado
en la utilización de enzimas.
De acuerdo con los estudios realizados por Rani et al 23 , y Kasonkanji et al 24 , acerca de la clasificación de leucemias
agudas utilizando esta técnica se hace uso de un panel de marcadores de incluye
CD34, MPO, CD117, CD3, CD20, PAX5, TdT y CD10. Para diferenciar entre LLA y LMA
se usaron MPO y CD117. En cuanto a la distinción en la línea linfoide, la LLA-B
fue positiva para CD34, CD20, PAX5, CD10, mientras que la LLA – T mostró
positividad para CD34, CD3 y TdT. También se menciona que otros antígenos de
células T como CD2, CD5 y CD7 no son específicos de este linaje y pueden manifestarse
de forma anormal en la LMA.
Citometría de flujo.
Esta técnica representa una tecnología potente capaz
de procesar decenas de miles de partículas por segundo y de identificar
subpoblaciones mediante herramientas de análisis multiparamétrico25 . Para realizar esta técnica las células o
partículas que se van a analizar deben encontrarse suspendidas en un fluido
como la solución salina tamponada con fosfato (PBS). Esta suspensión requiere
someter a la muestra, que en este caso es un aspirado de MO o sangre periférica
(únicamente en caso de que la MO sea de mala calidad o se observe una blastosis
periférica masiva), a un proceso de lisis – tinción – fijación y lavado. Las
células van a ser teñidas con un panel de anticuerpos conjugados a fluorocromos
como isotiocianato de fluoresceína (FITC), ficoeritrina (PE), aloficocianina
(APC), proteína peridinina clorofila cianina 5.5 (PerCP Cy5.5), APC H7 y PE
Cyanin 7 (PE Cy 7)26–28 .
Al pasar por el citómetro de flujo, las células se
disparan a través de los rayos láser a altas velocidades y es en esta
intersección en donde la óptica de recolección de luz capta la fluorescencia y
la dispersión de luz de cada célula25,26 . El uso de uno o varios láseres, al
incidir sobre las células, permite medir sus características por la dispersión
frontal, lo que refleja el tamaño y por la dispersión lateral la complejidad
interna o granularidad de cada célula. Además, al incidir el láser sobre las
moléculas de fluorescencia estas se excitan y envían una señal15,25 .

Figura 2. Pasos para el análisis de muestras mediante
citometría de flujo.
Es así como mediante estas técnicas se pueden detectar
los antígenos presentes en la superficie de las células blásticas de cada
estirpe (T o B). Además, también se identifican a los subtipos que van a
corresponder con los diferentes estadios de diferenciación de los linfocitos
normales15 .

Tabla 3. Clasificación de la LLA por inmunofenotipos. 15, 29
También cabe recalcar que enfrenta desafíos, como el
bajo volumen y la baja viabilidad celular, resultados engañosos debido al
cambio fenotípico, así como el manejo de grandes cantidades de datos, además de
que se considera que su sensibilidad es subóptima 21,29 . Es por ello por lo que se busca
implementar el uso del flujo de próxima generación, que ofrece mejorar la
interoperabilidad y sensibilidad29 .
En casos de fibrosis en la MO, la cantidad de células
obtenidas para CF disminuye considerablemente, lo que a veces impide su uso
para el diagnóstico, convirtiendo al estudio de la biopsia de MO mediante IHQ
en la herramienta diagnóstica alternativa21 .
Citogenética
La LLA se describe como una enfermedad de origen
genético, debido a que en la gran mayoría de casos se encuentran alteraciones
genéticas adquiridas (mutaciones somáticas), las cuales favorecen el incremento
del proceso proliferativo, la supervivencia prolongada y/o la diferenciación
incorrecta de las células progenitoras linfoides. Es por ello por lo que tanto
los hallazgos citogenéticos como los genéticos moleculares representan factores
pronósticos importantes al momento del diagnóstico de la LLA, ya sea en niños o
adultos30 .
Dentro de la citogenética se pueden mencionar
principalmente al análisis convencional o estándar y la hibridación in situ
por fluorescencia (FISH, por sus siglas en inglés).
·
Análisis
citogenético estándar.
En esta técnica se pueden detectar microscópicamente
las anomalías en los cromosomas, sin importar si son anormalidades en el número
de cromosomas o en su estructura31 . Para ello es necesario obtener células en
metafase, en donde las cromátidas se encuentran condensadas y la morfología de
los cromosomas está bien definida32 . Las muestras se someten a cultivos in
vivo no estimulados en un período corto (24 a 48 horas), y posterior a este
tiempo se detiene la división celular al exponer el cultivo a diversas
sustancias (colamida principalmente). Estas muestras se van a teñir usando
técnicas de banda (generalmente bandas G) y se analizan en el microscopio. Para
poder confirmar que un caso es citogenéticamente normal es necesario analizar
por lo menos 20 cariotipos de una muestra de MO y en caso de encontrar
aberraciones cromosómicas, estas se consideran relevantes cuando son clonales.
Esto significa que deben encontrarse en al menos dos células en metafase si se
trata de una aberración estructural idéntica o de una ganancia de un cromosoma
intacto (trisomía), en el caso de un cromosoma que falta (monosomía), deben
estar presentes en al menos tres células30,31 .

Figura 3. Protocolo para el análisis de cariotipo
convencional.
Mediante esta técnica se pueden detectar aneuploidías
(hiper o hipodiploidías) y otros reordenamientos cromosómicos comunes. Sin
embargo, el cariotipo convencional no suele ser lo suficientemente sensible en
la detección de reordenamientos que son crípticos, es decir, que son muy
pequeños y más complejos10 . Además, la precisión del análisis de
cariotipos depende de la obtención de metafases de alta calidad provenientes de
células neoplásicas. Sin embargo, en casos donde los blastos tienen un bajo
potencial de proliferación o en situaciones de alta hiperdiploidía, es posible
que no se generen metafases analizables, lo que podría llevar a una
interpretación errónea de los resultados32 .
En la LLA-B, se encuentran varias alteraciones como
diferentes tipo de ploidía: hiperdiploidía (> 50 cromosomas), hipodiploidía
(entre 24 y 31 cromosomas denominado casi hiploide o entre 32 y 39 cromosomas
denominado bajo haploide) y traslocaciones como t(9;22), KMT2A -r,
t(5;14); t(1;19) . Mientras que, en la LLA-T, aún no se ha establecido una
clasificación exacta acerca de las alteraciones detectadas mediante el
cariotipo convencional, lo cual puede deberse a que la cantidad de casos
citogenéticamente normales es mayor que en la LLA-B30,33 .
Aproximadamente un tercio de los pacientes con LLA-T
presenta una translocación que involucra alguno de los genes del receptor de
células T (TCR), con puntos de ruptura en 14q11 (TCRA/TCRD) o 7q34 (TCRB).
La translocación más frecuente en adultos es t(10;14)(q24;q11.2), la cual
provoca la sobreexpresión del gen TLX1 (HOX11) y está vinculada a un pronóstico
favorable. En niños, alrededor del 3 y el 6% de los casos presentan una
translocación t(1;14)(p32;q11.2), que coloca al gen TCRD junto al gen TAL1, lo
que provoca la sobreexpresión de este último. Además, esta sobreexpresión puede
ocurrir debido a una deleción críptica en el cromosoma 1, lo que provoca una
fusión entre los genes TAL1 y STIL. Esta fusión específica, TAL1-STIL, se
observa en un 16–26% de los casos, convirtiendo a las alteraciones relacionadas
con TAL1 (tanto la translocación como la fusión) en las anomalías genéticas más
comunes en este tipo de leucemia infantil30 .
Si bien el cariotipo convencional ofrece una
perspectiva amplia y completa de las aberraciones cromosómicas presenta ciertas
limitaciones como el realizar un cultivo celular que demanda mucho tiempo y que
tiene probabilidades de fracaso, una interpretación que depende de la
experiencia del analista y una resolución máxima de entre 5 a 7 megabases (Mbp)34 .
·
FISH
El principio en el cual se basa esta técnica es la
hibridación de sondas de ADN de cadena simple marcadas con fluoróforos a sus
secuencias genómicas complementarias32 . El procedimiento se puede iniciar con las
células preparadas en la técnica del cariotipo convencional, depositando las
mismas en un portaobjetos e incubando durante 2 minutos a 45°C en una placa
caliente, para luego empapar el portaobjetos en citrato salino de sodio (SSC)
por 5 minutos. Posteriormente, se somete a un proceso de deshidratación con
diferentes grados de alcohol (70%, 85% y 100%) y se procede a agregar la sonda,
se coloca un cubreobjetos y la muestra se somete a una desnaturalización seguido
de una hibridación durante toda la noche. Al día siguiente se realiza otra
hibridación con SSC y finalmente los portaobjetos se tiñen con los fluorocromos
que pueden ser 4,6-diamino-2-fenilindol (DAPI), isotiocianato de
fluoresceína (FITC), cloruro de ácido sulforodamina 101, entre otras. Las
señales de fluorescencia se van a detectar utilizando un microscopio específico
para este fin35,36 . Cabe destacar que las sondas que se
utilizan son fragmentos de ADNc o ADN genómico que son complementarias a
secuencias específicas en el genoma humano30 .

Figura 4. Protocolo para el procesamiento de muestras
mediante FISH.
La ventaja de la FISH es que no requiere de células
vivas, ya que se pueden analizar células tanto en metafase como en interfase, a
diferencia del cariotipo convencional que ocupa exclusivamente células en
metafase, además permite analizar con mayor rapidez un gran número de células e
identificar cambios estructurales que son más sutiles para la resolución de la
citogenética convencional37 . Sin embargo, no puede identificar
pequeñas deleciones y mutaciones38 .
Actualmente, esta técnica es usada en la detección de
translocaciones, inversiones, inserciones, microdeleciones y en la
identificación de cromosomas llamados marcadores o de referencia, además
también es útil en la delimitación de los puntos de ruptura a nivel cromosómico39 .
Se utilizan varios tipos de sondas que cumplen con una
función en específico de acuerdo con lo que se intenta encontrar o detectar.
Entre estas se encuentran las sondas centroméricas, sondas de pintado
cromosómico y las sondas de secuencia única o locus específico. Mediante esta
última sonda se puede detectar al cromosoma Filadelfia en la LLA, que se forma
debido a la traslocación t(9;22), presente en el 5% de los casos y que
representa un mal pronóstico37 . También se ha logrado detectar el gen de
fusión ETV6/RUNX1, identificar patrones de hibridación atípicos y la
amplificación intracromosómica del 21 (iAMP21)39 .
Técnicas moleculares
De acuerdo con la clasificación actualizada por la OMS
en el 2016, de todas las anomalías genéticas recurrentes que se presentan en la
LLA, las traslocaciones suelen ser las más frecuentes. Estas al ser analizadas
desde una perspectiva molecular son las causantes de la formación de genes de
fusión, creados a partir del intercambio de material entre cromosomas y que se
puede detectar en el ácido ribonucleico (ARN) mediante la reacción en cadena de
la polimerasa con retrotranscripción (RT-PCR)40 .
-
RT-PCR
Se describe como una variante de la PCR convencional y
consta de 2 procesos41 . El primero de ellos inicia con la
trasncripción inversa, en donde se va a tomar un fragmento de ARN para
convertirlo en ADN complementario (ADNc) por acción de la trasncriptasa
inversa. El ADNc obtenido va a servir como molde para realizar la PCR convencional,
que inicia con la desnaturalización del ADN para separar las cadenas dobles,
después se añaden los cebadores o primers para su unión con las cadenas simples
de ADN que, cabe destacar, van a ser diseñadas exclusivamente para las
necesidades de cada estudio realizado. Por último, la elongación o extensión
que es catalizada por la ADN polimerasa, resultando en la duplicación del
material genético14,41,42 .

Figura 5. Pasos esenciales en la RT-PCR.
Presenta una alta sensibilidad (0,01% a 0,001%),
superior a otras técnicas como la CF, que le permite detectar una célula
maligna entre 10 000 a 100 000 células normales14 . Utilizando cebadores dirigidos hacia
distintos genes de fusión se han logrado detectar varios reordenamientos
importantes como BCR::ABL1, TCF3::PBX1 , ETV6::RUNX1 y UBTF::ATXN7L3.
Además, se considera que la implementación de esta metodología es
especialmente útil para la detección de reordenamientos crípticos y
comprobación de la expresión y estructura de nuevos genes de fusión
identificados por otras metodologías como FISH. Sin embargo, su uso se limitado
por la gran heterogeneidad molecular que generan las fusiones génicas, tal es
el caso del subtipo BCR::ABL1-like, en el que están involucradas varias
quinasas 10 .
Secuenciación de nueva generación
Se considera que, aún en la actualidad al momento de
realizar el análisis genómico de la LLA se utilizan técnicas de bajo
rendimiento como la FISH, debido a que el número de alteraciones que detecta es
muy limitado, es por ello por lo que se espera modernizar las técnicas
diagnósticas con la introducción de otras tecnologías como la secuenciación de
nueva generación (NGS, por sus siglas en inglés)43 . También se puede utilizar para monitorear
la EMR, consiguiendo mejores resultados al alcanzar una sensibilidad de hasta
1x10-6, de acuerdo con el estudio realizado por Short et al 44 , del total de pacientes que tuvieron
resultados negativos para EMR mediante CF, el 46% resultó positivo al realizar
el estudio utilizando NGS. Se demostró de esta manera que, esta técnica puede
detectar incluso aquellos casos en los que las células leucémicas son casi
imperceptibles para otras técnicas que se usan comúnmente en el diagnóstico
convencional de la LLA.
La NGS es una técnica avanzada que permite identificar
de manera eficiente y rápida cambios moleculares en las células tumorales.
Tradicionalmente estos cambios genéticos se detectaban mediante citogenética
convencional, FISH o PCR, sin embargo, al buscar mejorar la relación
coste-eficiencia se implementó esta nueva técnica capaz de detectar múltiples
aberraciones genéticas a la vez45 . De acuerdo con la complejidad que
represente el análisis requerido y la información que se quiera obtener se
pueden realizar distintos tipos de secuenciación, entre las que se incluye a la
secuenciación del genoma completo (WGS), secuenciación del transcriptoma
(RNA-seq) y secuenciación del exoma completo (WES)43 . Cabe señalar que todas estas técnicas no
forman parte del estudio estándar para el diagnóstico de la LLA, sin embargo,
se llevan a cabo en los laboratorios de investigación para lograr establecer
una clasificación mejor definida de esta enfermedad29 .
En un principio esta técnica se veía limitada tanto
por su precio como por la complejidad de su uso. A partir de 2001, cuando se
estaban realizando los primeros experimentos el costo se ha reducido
drásticamente de 100 millones de dólares a 1000 dólares, aunque este precio aún
es considerablemente alto se espera que siga bajando en los próximos años.
Asimismo, las herramientas bioinformáticas empleadas para analizar la gran
cantidad de datos obtenidos a partir de la secuenciación han sido mejoradas
mediante el perfeccionamiento de nuevos algoritmos y canales43 .
WGS
Esta técnica se divide en 4 procesos esenciales:
preparación de la muestra, preparación de la biblioteca, generación de grupos y
secuenciación46,47 . El ADN de alto peso molecular se aísla a
partir de sangre completa (EDTA o citrato), tejidos extirpados quirúrgicamente
o biopsias, para luego ser fragmentado y generar la biblioteca. Aquí también se
agregan los adaptadores y el ADN enlazador. Posteriormente, las bibliotecas se
cargan en una celda de flujo y se sitúan en un secuenciador en donde los
fragmentos de ADN van a amplificarse mediante una polimerasa. Con un detector
láser de fluorescencia se detecta la señal producida y los datos finalmente se
transfieren a una computadora de alto rendimiento para ser analizados y
comparados con el genoma de referencia humano43,46 . Por lo cual, esta técnica es
especialmente valiosa para estudiar o detectar variaciones estructurales43 .

Figura 6. Protocolo para análisis del genoma y exoma
completo.
WES
Considerada útil para estudiar mutaciones puntuales,
especialmente en subclones leucémicos durante la recaída. Esta técnica comparte
un flujo de trabajo bastante similar a WGS, con la excepción de que, a
diferencia de dicha técnica, esta va a centrarse únicamente en las regiones
codificantes, es decir, los exomas. Cabe mencionar que se van a ocupar bases de
datos como la del Consorcio de Agregación de Exomas para obtener más
información del efecto biológico de las alteraciones identificadas43 .
RNA-seq
Se emplea para analizar la expresión de ARNm o ARN no
codificante, y puede identificar tanto mutaciones en la secuencia como genes de
fusión, los cuales también pueden ser detectados por el WGS43 . Este análisis consta de 3 pasos
fundamentales: alineamiento, cuantificación y análisis diferencial48,49 . El proceso de preparación de una
biblioteca de RNA-seq comienza con la obtención y aislamiento del ARN de una
muestra biológica, que puede provenir de una línea celular o de un tejido
previamente congelado. Posteriormente, el ARN es transformado en ADNc mediante
una transcripción inversa, el ADN obtenido va a ser fragmentado en secuencias
cortas. La biblioteca creada a partir de este proceso se convierte en la
entrada a una plataforma de secuenciación que va a generar las lecturas
correspondientes al comparar o alinear estas con otra secuencia de referencia50 .

Figura 7. Protocolo para el análisis del transcriptoma
completo.

Tabla 4. Alteraciones detectadas en varios estudios
por distintas técnicas.

Tabla 5. Principales beneficios y limitaciones de las diferentes
técnicas analizadas.
Desafíos en países en vías de desarrollo
Se recalca ampliamente que las tasas de curación y
supervivencia de la LLA rondan el 90%, sin embargo, estos datos pertenecen
únicamente a los países de altos ingresos. Esto se debe a que las técnicas más
avanzadas ya han sido implementadas en dichos lugares52 . En contraste, tanto en América Latina
como en otros países de ingresos medios bajos, los resultados están
significativamente por debajo de los estándares internacionales establecidos,
tanto en pacientes pediátricos como adultos. Además, la disponibilidad de las
pruebas basadas en la citogenética o biología molecular varía ampliamente y
depende en gran medida del ámbito económico.
Actualmente, la medición de la EMR es considerado como
el factor pronóstico independiente más importante, ya que, facilita la
adaptación de la intensidad de la quimioterapia y el ajuste de las estrategias
de consolidación7 . Los ensayos preferidos para realizar estos
análisis son la PCR y la CF, está última siendo la menos accesible y asequible,
debido a que la distribución de laboratorios que realicen técnicas genéticas y
citogenéticas se concentra por lo general en áreas urbanas principales, lo que
provoca una alta demanda que colapsa los sistemas de salud en Latinoamérica. Lo
cual subraya la necesidad de mejoras en la gestión gubernamental que permita el
acceso a estas herramientas.
La implementación de técnicas más avanzadas es de suma
importancia debido a que mediante la detección de anomalías genéticas
específicas que son recurrentes y la medición de la EMR al finalizar la terapia
de inducción es posible establecer una clasificación de riesgo (bajo, estándar,
alto o muy alto). Esta combinación ha dado grandes resultados en cuanto a la
mejora en las tasas de supervivencia en los últimos 30 años 7,53 . Además, se necesitan encontrar nuevos
enfoques terapéuticos para los casos de LLA de alto riesgo, cuyo pronóstico
sigue siendo desfavorable aún con la aplicación de una terapia intensiva. Para
ello es necesario conocer con mucha más profundidad las alteraciones
moleculares que son responsables de la resistencia a estos tratamientos 53 .

Tabla 6. Principales alteraciones y/o mutaciones
detectadas por cada una de las técnicas.
La integración de múltiples técnicas diagnósticas,
desde los métodos convencionales hasta las tecnologías más avanzadas, ha
revolucionado el abordaje clínico de la Leucemia Linfoide Aguda (LLA). Si bien
las técnicas tradicionales como el hemograma, el análisis morfológico y las
tinciones citoquímicas siguen siendo fundamentales en el diagnóstico inicial,
su sensibilidad y especificidad han sido complementadas por herramientas más
precisas, como la citometría de flujo, el cariotipo convencional y la
hibridación in situ por fluorescencia (FISH). A su vez, los enfoques
moleculares como RT-PCR y la secuenciación de nueva generación (NGS) han
permitido detectar con mayor precisión las alteraciones genéticas y los
subtipos de riesgo, facilitando así la estratificación del tratamiento y la
medición de la enfermedad mínima residual (EMR). Estos avances no solo han
incrementado las tasas de supervivencia, especialmente en países desarrollados,
sino que también subrayan la necesidad urgente de implementar dichas
tecnologías en países en vías de desarrollo para reducir la brecha existente en
el acceso a un diagnóstico preciso y oportuno.
CONCLUSIONES
Los avances en las técnicas de diagnóstico de la LLA
han revolucionado el enfoque clínico de esta enfermedad, permitiendo una
identificación más precisa y rápida de los subtipos moleculares y alteraciones
genéticas, consideradas como las más cruciales en cuanto a la elección de
tratamiento y la estratificación de riesgo, ya que partiendo de estas
alteraciones se establece un peor o mejor pronóstico. Las técnicas
tradicionales como el hemograma y el frotis sanguíneo aún cumplen con un papel
importante en el diagnóstico presuntivo de esta enfermedad, ya que, estos
resultados son el punto de partida para análisis más exhaustivos. Asimismo, las
tinciones citoquímicas también representaron una parte fundamental en el
diagnóstico, especialmente al momento de diferenciar la LLA de la LMA, aunque
con el tiempo su uso fue desplazado hasta quedar prácticamente obsoletas.
No obstante, estas técnicas han sido complementadas
y/o superadas en especificidad y sensibilidad por tecnologías más avanzadas,
tales como la IHQ, que permite identificar marcadores específicos en las
células; la CF, utilizada para clasificar subtipos celulares; el cariotipo
convencional y FISH, que detectan alteraciones cromosómicas; y la RT-PCR, que
permite identificar translocaciones (genes de fusión) y otras mutaciones con
gran precisión a nivel molecular. Al integrar todas estas técnicas en un solo
flujo se logra una gran mejora en la precisión diagnóstica y también facilita
la estratificación de riesgos, la implementación de tratamientos mucho más
específicos y el monitoreo de la evolución de la enfermedad mediante el estudio
de la EMR.
Aunque las tasas de curación y supervivencia han
incrementado en los últimos años, esto ha ocurrido principalmente en países de
ingresos altos, quedando en evidencia la brecha tecnológica existente con los
países en vías de desarrollo. Es imprescindible garantizar un acceso equitativo
a métodos diagnósticos más avanzados para mejorar los resultados clínicos,
especialmente en adultos, que suele ser la población con los peores
pronósticos. Además, la investigación en torno a las alteraciones moleculares responsables
de la resistencia al tratamiento es esencial para desarrollar terapias más
efectivas mediante la clasificación de riesgo, particularmente para los
pacientes con LLA de alto riesgo, cuyo pronóstico sigue siendo desfavorable.
Técnicas como la NGS no han sido
implementadas en todos los laboratorios o bien están en vías de adaptación y
desarrollo para que sean más accesibles, ya sea por la complejidad que
representan estos análisis y por lo tanto el personal altamente especializado
que se requiere para su ejecución, o bien por los altos costos que representa
el realizar uno de estos estudios.
Financiación: Este artículo no
recibió financiación externa.
Conflicto de
intereses: Los autores
declaran no tener ningún conflicto de intereses.
REFERENCIAS
1. Brown PA, Shah B, Advani A, Aoun P,
Boyer MW, Burke PW, et al. Acute Lymphoblastic Leukemia, Version 2.2021, NCCN
Clinical Practice Guidelines in Oncology. Journal of the National
Comprehensive Cancer Network. 2021;19(9).
2. Elsayed B, Elhadary M, Elshoeibi RM,
Elshoeibi AM, Badr A, Metwally O, et al. Deep learning enhances acute
lymphoblastic leukemia diagnosis and classification using bone marrow images. Vol. 13, Frontiers in Oncology. 2023.
3. José
M, Rodríguez M, De La Paz K, Salinas A, Alexander K, Cepeda S, et al. Leucemia
linfoblástica aguda diagnostico. RECIMUNDO: Revista Científica de la
Investigación y el Conocimiento, ISSN-e 2588-073X, Vol 4, No 2,
2020, págs 53-63. 2020;4(2).
4. Mejia-Arangure JM, McNally RJQ,
Pombo-de-Oliveira MS. Editorial: Prevention in Acute Leukemias in Children.
Vol. 9, Frontiers in Public Health. Frontiers Media S.A.; 2021.
5. Kansal R. Diagnosis and Molecular
Pathology of Lymphoblastic Leukemias and Lymphomas in the Era of Genomics and
Precision Medicine: Historical Evolution and Current Concepts—Part 2:
B-/T-Cell Acute Lymphoblastic Leukemias. Lymphatics. 2023;1(2).
6. Dong Y, Shi O, Zeng Q, Lu X, Wang W, Li
Y, et al. Leukemia incidence trends at the global, regional, and national
level between 1990 and 2017. Exp Hematol Oncol.
2020 Dec 19;9(1):14.
7. Colunga-Pedraza
PR, Colunga-Pedraza JE, Peña-Lozano SP, Gómez-De León A, Ruiz-Delgado GJ,
Ribeiro RC. Diagnosis and
treatment of acute lymphoblastic leukemia in Latin America. Hematology. 2022
Dec 31;27(1):971–6.
8. Sharma N, Gupta R, Kotru M, Gomber
Sunil,, Vardhan Gautam H. Higher baseline natural killer cell counts are
associated with a lower 8-day blast count and lower day 33 minimal residual
disease in children with pediatric B-acute lymphoblastic leukemia. Am J Blood Res. 2023;13(1):53–60.
9. Farias
MG, Castro SM de. Diagnóstico laboratorial das leucemias linfóides agudas. J Bras Patol
Med Lab. 2004;40(2).
10. Duffield AS, Mullighan CG, Borowitz MJ.
International Consensus Classification of acute lymphoblastic
leukemia/lymphoma. Vol. 482, Virchows Archiv. 2023.
11. Shafique S, Tehsin S. Computer-Aided
Diagnosis of Acute Lymphoblastic Leukaemia. Comput Math Methods Med.
2018;2018:1–13.
12. Pui CH, Nichols KE, Yang JJ. Somatic and
germline genomics in paediatric acute lymphoblastic leukaemia. Vol. 16, Nature
Reviews Clinical Oncology. Nature Publishing Group; 2019. p. 227–40.
13. González Llano O. The complete blood
count in the early diagnosis of acute leukemia in children. Medicina Universitaria. 2016;18(73).
14. Jaime
Pérez JC, Gómez Almaguer D. Hematología, la sangre y sus enfermedades.
(Spanish). Vol. 7, Medicina Universitaria. 2005.
15. Moraleda
JJ. Pregrado de Hematología. Vol. 4, Suparyanto dan Rosad (2015. 2017.
16. Aldama
D, Sotomayor G. Sensibilidad y especificidad de morfología, inmunofenotipo e
histopatología para el diagnóstico de leucemias agudas en un centro del
noreste de México. Revista Hematología. 2023;27(2).
17. Castañeda-Huerta
JE. Leucemia linfoblástica aguda. Revista Médica MD. 2009;1(4).
18. Santos
J da S, Sá LFP de, Mata JP de O, Teixeira A da S, Crisanto AVS de MS, Amorim
GR, et al. Diagnóstico da
leucemia linfoblástica aguda em crianças. Research, Society and Development.
2022 Jul 13;11(9):e39411919078.
19. Gonzáles Cruz E de J, Díaz Contreras A,
Gómez Aburto DH, Rivera Rosado FE, de la Cruz Nicolás MÁ. Manual de tinciones
citoquímicas especiales en hematología. CECan; 2019.
20. Olsen RJ, Chang CC, Herrick JL, Zu Y,
Ehsan A. Acute leukemia immunohistochemistry: A systematic diagnostic
approach. Vol. 132, Archives of Pathology and Laboratory Medicine. 2008.
21. ÖZDEMİR Ç, DÜZENLİ KAR Y, EROĞLU N, ŞENOL
Y, EKER İ, ŞAHİN M. Evaluation of the Correlation of Immunohistochemical
Findings with Flow Cytometric Findings in Newly Diagnosed Pediatric Acute
Lymphoblastic Leukemia Patients. Journal of Contemporary Medicine. 2023 Mar
22;13(2):318–25.
22. Magaki S, Hojat SA, Wei B, So A, Yong WH.
An Introduction to the Performance of Immunohistochemistry. In 2019. p.
289–98.
23. Rani HS, Hui M, Uppin MS, Uppin SG,
Sadashivudu G, Paul TR. Utility of Immunohistochemistry on Bone Marrow
Trephine Biopsy for the Diagnosis and Classification of Acute Leukemia. INDIAN
JOURNAL OF MEDICAL AND PAEDIATRIC ONCOLOGY. 2020 Oct;41(5):683–7.
24. Kasonkanji E, Kimani S, Skiver B, Ellis
G, Seguin R, Kaimila B, et al. Clinical Characteristics and Outcomes of Acute
Lymphoblastic Leukemia in Adolescents and Young Adults in Malawi. JCO Glob
Oncol. 2022;(8).
25. Robinson JP. Flow cytometry: past and
future. Vol. 72, BioTechniques. 2022.
26. Cossarizza A, Chang H, Radbruch A, Acs A,
Adam D, Adam‐Klages S, et al. Guidelines for the use of flow cytometry and
cell sorting in immunological studies (second edition). Eur J Immunol. 2019
Oct 21;49(10):1457–973.
27. Dworzak MN, Buldini B, Gaipa G, Ratei R,
Hrusak O, Luria D, et al. AIEOP ‐ BFM Consensus Guidelines 2016 for Flow
Cytometric Immunophenotyping of Pediatric Acute Lymphoblastic Leukemia.
Cytometry B Clin Cytom. 2018 Jan 21;94(1):82–93.
28. Pavithra P, Laxminarayana SLK, Manohar C,
Belurkar S, Kairanna N V. Transition from morphologic diagnosis to
immunophenotypic diagnosis of acute leukemia-experience of establishing a new
flow cytometry laboratory. J Hematop. 2019 Oct;12(4):191–9.
29. Gökbuget N, Boissel N, Chiaretti S,
Dombret H, Doubek M, Fielding A, et al. Diagnosis, prognostic factors, and
assessment of ALL in adults: 2024 ELN recommendations from a European expert
panel. Vol. 143, Blood. 2024.
30. Mrózek K, Harper DP, Aplan PD.
Cytogenetics and Molecular Genetics of Acute Lymphoblastic Leukemia. Vol. 23,
Hematology/Oncology Clinics of North America. 2009.
31. Mrózek K, Carroll AJ, Maharry K, Rao KW,
Patil SR, Pettenati MJ, et al. Central review of cytogenetics is necessary for
cooperative group correlative and clinical studies of adult acute leukemia:
the Cancer and Leukemia Group B experience. Int J Oncol. 2008
Aug;33(2):239–44.
32. Dhabe A, Islam R, Ramakrishnan K, Parihar
M. Role of Cytogenetics and FISH in Laboratory Workup of B Cell Precursor
Acute Lymphoblastic Leukemia. Indian Journal of Medical and Paediatric
Oncology. 2023 Oct 17;44(05):482–93.
33. Genescà E, Morgades M, González-Gil C,
Fuster-Tormo F, Haferlach C, Meggendorfer M, et al. Adverse prognostic impact
of complex karyotype (≥3 cytogenetic alterations) in adult T-cell acute
lymphoblastic leukemia (T-ALL). Leuk Res. 2021 Oct;109:106612.
34. Mareschal S, Palau A, Lindberg J, Ruminy
P, Nilsson C, Bengtzén S, et al. Challenging conventional karyotyping by
next-generation karyotyping in 281 intensively treated patients with AML.
Blood Adv. 2021 Feb 23;5(4):1003–16.
35. Kulkarni N V, Shetty V V, Prasad HL K,
Arumugam M, Krishna R, Shetty RA, et al. Evaluation of the cytogenetic profile
in patients with acute leukaemia. Polish Journal of Pathology.
2023;74(2):89–97.
36. Yadav V, Raveendranath V, Ganesan P, Kar
R, R P, Manivannan P. Immunophenotypic Characteristics and Cytogenetic
Analysis of Adolescent and Young Adult B-Cell Acute Lymphoblastic Leukemia:
Correlations With Clinicopathological Parameters. Cureus. 2024 Sep 5;
37. Lavaut Sánchez K, Hernández Aguilar N,
Ruiz Moleón V. Hibridación in situ fluorescente: Herramienta en el diagnóstico
de las hemopatías malignas. Revista Cubana de Hematologia, Inmunologia y
Hemoterapia. 2016;32(1).
38. Ibargoyen-Roteta N, Gutiérrez-Ibarluzea
I, Garcia Ruiz JC, Lawry C, Urruticoechea Ribate A. Secuenciación masiva en el
diagnóstico de las leucemias agudas. 2023.
39. González García S, Lavaut Sánchez K.
Técnicas de citogenética para el estudio de las leucemias. Revista Cubana de
Hematología, Inmunología y Hemoterapia. 2022;38(2).
40. Huguet F, Récher C. Leucemia
linfoblástica aguda. EMC - Tratado de Medicina. 2024 Mar;28(1):1–6.
41. Díaz-Alonso C, Garrote-Santana H, Ana
maría amor-Vigil C, Suárez-González Y, González-Mugica Romero R.
Cuantificación de ácido ribonucleico para la realización de la técnica de RT-
PCR. Revista Cubana de Hematologia, Inmunologia y Hemoterapia. 2013;29(3).
42. Okuku P, Kwenda G, Samutela M, Nkhoma P,
Mantina H. Molecular detection of fusion oncogenes in zambian patients with
acute lymphoblastic leukemia. Int J Appl Basic Med Res. 2020;10(4).
43. Coccaro N, Anelli L, Zagaria A, Specchia
G, Albano F. Next-Generation Sequencing in Acute Lymphoblastic Leukemia. Int J
Mol Sci. 2019 Jun 15;20(12):2929.
44. Short NJ, Kantarjian H, Ravandi F,
Konopleva M, Jain N, Kanagal-Shamanna R, et al. High-sensitivity
next-generation sequencing MRD assessment in ALL identifies patients at very
low risk of relapse. Blood Adv. 2022 Jul 12;6(13):4006–14.
45. Cho YU. The role of next-generation
sequencing in hematologic malignancies. Vol. 59, Blood Research. 2024.
46. Bagger FO, Borgwardt L, Jespersen AS,
Hansen AR, Bertelsen B, Kodama M, et al. Whole genome sequencing in clinical
practice. BMC Med Genomics. 2024 Jan 29;17(1):39.
47. Brlek P, Bulić L, Bračić M, Projić P,
Škaro V, Shah N, et al. Implementing Whole Genome Sequencing (WGS) in Clinical
Practice: Advantages, Challenges, and Future Perspectives. Vol. 13, Cells.
2024.
48. Jiang G, Zheng JY, Ren SN, Yin W, Xia X,
Li Y, et al. A comprehensive workflow for optimizing RNA-seq data analysis.
BMC Genomics. 2024 Jun 24;25(1):631.
49. Spinozzi G, Tini V, Adorni A, Falini B,
Martelli MP. ARPIR: automatic RNA-Seq pipelines with interactive report. BMC
Bioinformatics. 2020;21.
50. Deshpande D, Chhugani K, Chang Y,
Karlsberg A, Loeffler C, Zhang J, et al. RNA-seq data science: From raw data
to effective interpretation. Front Genet. 2023 Mar 13;14.
51. Brown LM, Lonsdale A, Zhu A, Davidson NM,
Schmidt B, Hawkins A, et al. The application of RNA sequencing for the
diagnosis and genomic classification of pediatric acute lymphoblastic
leukemia. Blood Adv. 2020;4(5).
52. Inaba H, Pui CH. Advances in the
diagnosis and treatment of pediatric acute lymphoblastic leukemia. J Clin Med.
2021;10(9).
53. Harvey RC, Mullighan CG, Chen IM, Wharton
W, Mikhail FM, Carroll AJ, et al. Rearrangement of CRLF2 is associated with
mutation of JAK kinases, alteration of IKZF1, Hispanic/Latino ethnicity, and a
poor outcome in pediatric B-progenitor acute lymphoblastic leukemia. Blood.
2010 Jul 1;115(26):5312–21.
Received: November 29, 2024 /
Accepted: December 15, 2025 / Published: March 15,
2025
Citation: Pico-Sánchez D F, Rosero-Freire D A. De la
microscopía a la secuenciación genética: La evolución en las técnicas de
diagnóstico de la Leucemia Linfoide Aguda. Bionatura
Journal. Bionatura Journal. 2025;2 (1):11. doi: 10.70099/BJ/2025.02.01.11
Additional information Correspondence should be addressed to da.roserof@uta.edu.ec
Peer review information. Bionatura thanks anonymous reviewer(s) for their
contribution to the peer review of this work using https://reviewerlocator.webofscience.com/
ISSN.3020-7886
All articles published by Bionatura Journal are made
freely and permanently accessible online immediately upon publication, without
subscription charges or registration barriers.
Publisher's Note: Bionatura Journal stays neutral concerning
jurisdictional claims in published maps and institutional affiliations.
Copyright: © 2024 by the authors. They were submitted for possible open-access
publication under the terms and conditions of the Creative Commons Attribution
(CC BY) license (https://creativecommons.org/licenses/by/4.0/).




